Química
Determinación del peso molecular de una proteína
PRÁCTICA 1:
DETERMINACIÓN DEL PESO MOLECULAR DE UNA PROTEÍNA MEDIANTE ELECTROFORESIS EN GELES DE POLIACRILAMIDAS-SDS.
El objetivo es poder determinar el peso molecular de proteínas cuando se someten a un campo eléctrico y se hacen pasar a través de una matriz de tamaño de poro conocido (gel de poliacrilamida).
La formación del gel se realiza siguiendo la siguiente tabla, donde gracias a la solución de Acrimilamida obtenemos el entramado matriz. Dicho entramado se obtiene por la utilización del catalizador TEMED; que inicialmente está inactivo y es activado por el persulfato amónico.
| Gel Separador 15% | Gel Compactador 4% | |
| Sol. Acrilamida 30% | 10 ml | 2ml |
| 1,5 M Tris pH 8,8 | 5ml | - |
| 0.5 M Tris pH 6.8 | - | 2.5ml |
| DdH20 | 4.2ml | 5ml |
| 10% SDS | 200 (micro litros) | 100(micro litros) |
| Vacio durante 5 min. | ||
| 10% APS | 200 (micro litros) | 100 (micro litros) |
| TEMED | 40 (micro litros) | 25 (micro litros) |
PREPARACIÓN DE LAS MUESTRAS.
| 1 | 2 | 3 | 4 | 5 | |
| Tampón + (2-ME x 4) | - | 4 micro litros | - | 4 micro litros | - |
| Tampón - (2-ME x 4) | - | 4 micro litros | - | 4 micro litros | |
| Muestra 1 (15 micro g) | - | 12 micro litros | 12 micro litros | - | -- |
| Muestra 2 (15 micro g) | - | - | - | 12 micro litros | 12 micro litros |
| Patrones | 10 micro litros | - | - | - | - |
DESARROLLO DE LA ELECTROFORESIS.
1- Una vez obtenido el gel solidificado con sus pocillos, lavamos éstos con agua estéril. Colocamos el gel en la cubeta, rellenando el tanque y los pocillos con Tampón de Desarrollo.
2- Colocamos la muestra en el pocillo utilizando una microjeringa.
3- Desarrollamos la electroforesis durante una hora a 20 mA y a 20 ºC, para seguir la electroforesis utilizamos un marcador coloreado(azul de bromofenol)
TINCIÓN.
Retiramos el gel del sandwich y lo llevamos a una cubeta que contiene una solución de teñido. La tinción se realiza durante 30 min. a 50 ºC.
El gel se destiñe en otra solución durante 1 hora a 50 ºC.
DETERMINACIÓN DEL PESO MOLECULAR.
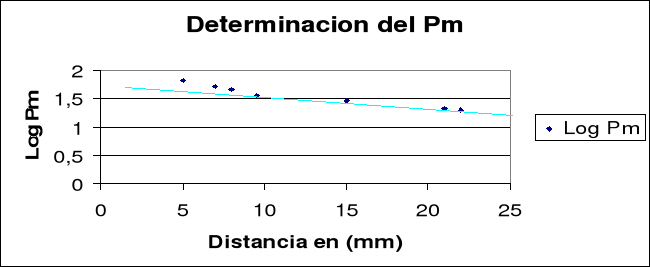
Medimos la movilidad electroforética de la proteína en mm. Esta distancia se toma desde el pocillo hasta la banda longitudinal del gel. Sabiendo que la distancia es inversamente proporcional al logaritmo de su peso molecular, realizamos una recta patrón con las proteínas de peso molecular conocido.
Los datos obtenidos en las mediciones quedan plasmados en la siguiente tabla, y representados en la correspondiente gráfica:
| PM (KD) | Log PM | mm | |
| BSA | 68 | 1.831 | 5 |
| GDH | 54 | 1.73 | 7 |
| ADH | 37 | 1.56 | 9.5 |
| Anh. Carbonica | 29 | 1.46 | 15 |
| Inh. Tripsina | 20 | 1.30 | 22 |
| Muetra 1 - 2-ME | 59.66 | 1.775 | 5 |
| Muetra 1 + 2-ME | 36.61 | 1.563 | 15 |
| Muetra 2 - 2-ME | 27.84 | 1.44 | 16 |
| Muetra 2 - 2-ME |
ASPECTOS A TENER EN CUENTA.
-El Azul de Bromofenol es un compuesto de muy bajo peso molecular en comparación con el de las proteínas, por esto avanza más rápido por el gel y no influye en la determinación del Pm de las proteínas.
-La electroforesis la detenemos cuando el Azul de Bromofenol llega al borde inferior del gel.
PRÁCTICA 2 :
GEL DE AGAROSA.
El objetivo es identificar y purificar fragmentos de DNA .
Las moléculas de DNA migran a través de una matriz de gel de agarosa de tamaño de poro conocido desde el polo positivo al polo negativo en función de su peso molecular en presencia de un campo eléctrico.
CARACTERÍSTICAS DEL GEL:
-El rango de separción de este gel oscila entre 100 pb y 60 Kb.
-El tamaño de poro depende del porcentaje de agarosa utilizado en la formación del gel.
DESARROLLO DE LA ELECTROFORESIS.
Con la utilización de un peine obtenemos unos pocillos sobre los cuales vamos a introducir la muestra.
Rellenamos los pocillos y los dos tanques de la cubeta con disolución tampón.
A continuación conectamos la cubeta a la fuente de alimentación de tal manera que en el polo negativo estén situados los pocillos del gel y el polo positivo al final del gel. Después de pinchar las muestras lo dejamos correr durante dos horas a 70 V.
Transferimos el gel a un recipiente donde lo teñimos con una disolución tampón+ Bromuro de Etidio, la cual nos permite la visualización de los fragmentos de DNA con luz UV (254 nm.).
DETERMINACIÓN DEL PESO MOLECULAR.
Medimos las distancias recorridas por los fragmentos de DNA en mm y como sabemos que la movilidad electroforética es inversamente proporcional al logaritmo del número de pares de bases de los fragmentos de DNA podemos elaborar la siguiente tabla:
| Pm | Log Pm | Distancias (mm) | |
| P1 | 21.2 | 1.32 | 11 |
| P2 | 5.1 | 0.7 | 19 |
| P3 | 4.9 | 0.69 | 20 |
| P4 | 4.3 | 0.63 | 22 |
| P5 | 3.5 | 0.54 | 29 |
| P6 | 2.0 | 0.3 | 30 |
| P7 | 1.9 | 0.28 | 32 |
| P8 | 1.6 | 0.2 | 34 |
| P9 | 1.4 | 0.156 | 38 |
| P10 | 0.95 | -0.022 | 40 |
| P11 | 0.83 | -0.08 | 42 |
| M1 | 4.58 | 0.661 | 21 |
| M2 | 1.88 | 0.276 | 32 |
| M3 | 0.9 | -0.039 | 41 |
PRATICA 3
DETERMINACIÓN ENZIMÁTICA DEL CONTENIDO EN GLUCOSA EN LOS MOSTOS COMERCIALES.
Las soluciones que vamos a utilizar en esta práctica son :
1-Tampón /MgSO4
2-ATP 150mM /NADP 12mM
3-Äcido perclórico 0,33M
4-Mezcla de enzimas
5-KOH 3 M / Imidazol 0,5 M
6- Glucosa 5 M
PREPARACIÓN DE LA MUESTRA
Se toma 1 ml de zumo en un tubo eppendorf, y se cetrifuga a 1300 rpm durante 10 min; para poder eliminar las partículas en suspensión. Después se recupera el sobrenadante y se reparte unos 500 microlitros en dos tubos. A continuación para poder desproteinizar las disoluciones añadimos 1 ml de Pca 0,33 N a cada una y volvemos a centrifugar en las mismas condiciones que antes. Recogemos el sobrenadante y lo neutralizamos añadiendo la solución KOH / Imidazol.
RECTA PATRÓN
La reacción enzimática tendrá lugar en las cubetas donde hemos pipeteados todos los componentes y mediante el espectrofotómetro determinaremos la absorbancia en cada una de las disoluciones.
Las absorbancias medidas en las muestras y en los patrones son:
| Absorbancias (Y) | Concentracion (X) | |
| Blanco | - | - |
| P1 | 0.628 | 0.166 |
| P2 | 0.517 | 0.125 |
| P3 | 0.1 | 0.083 |
| P4 | 0.082 | 0.042 |
| P5 | -0.019 | 0.021 |
| P6 | -0.0108 | 0.010 |
| M1 | 0.412 | 0.01 |
| M2 | 0.143 | 0.005 |
| M3 | 0.074 | 0.0025 |
Representamos graficamente los datos de la Absorbancia con la concentración de los patrones con el fin de obtener una recta. Con la cual podamos determinar la concentracion de las mustras.
| Concentracion (X) | Absorbancias (Y) | |
| P1 | 0.166 | 0.628 |
| P2 | 0.125 | 0.517 |
| P3 | 0.083 | 0.1 |
| P4 | 0.042 | 0.082 |
| P5 | 0.021 | -0.019 |
| P6 | 0.01 | -0.0108 |
| 0.15 | 0.5837 | |
| 0 | -0.085 | |
| M1 | 0.01 | 0.412 |
| M2 | 0.005 | 0.143 |
| M3 | 0.0025 | 0.074 |
| La recta de ajuste es: Y = 4,458 X - 0.085 | ||
Ahora con los valores de la absorbancia de las muestras y la recta de ajuste realizada obtendremos los valores de las concentraciones.
La recta de ajuste es: Y = 4.458 X - 0.085
Determinacion de la concentración 1:
Determinación de la Concentración 2:
Determinación de la Concentración 3:
La Media es:
PRACTICA 4
VALORACION ENZIMATICA
Las soluciones que vamos a utilizar son:
1-Tampón
2-Acido perclórico 0,33 N
3-50 mg de NAD 12mM disueltos en 3 ml de agua destilada
4-Patrones de etanol de 100 mM, 50 mM, 25 mM, 12 mM, 6 mM, y 3 mM
5-KOH 3 M, Imidazol 0,5 M
PREPARACION DE LA MUESTRA
Tomamos una muestra de 200 microlitros de vino y la centrifugamos a 1300 rpm durante 5 min,y luego recogemos unos 100 microlitros de sobrenadante. A continuación añadimos 1 ml de ácido perclórico para desproteinizar la disolución y después de mezclarlo bien en el vortex ya está preparado para centrifugarlo por segunda vez en las mismas condiciones que antes. Se recoge el sobrenadante y se neutraliza con la disolución número 5.
REACCION:
La reacción enzimática se realiza en las cubetas después de haber añadido los componentes.Llevamos éstas al espectrofotómetro para medir los distintos valores de absorbancias y así representar la recta patrón. Luego se determina la concentración del etanol en las muestras interpolando los valores obtenidos.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | |
| Tampon | 3ml | 3ml | 3ml | 3ml | 3ml | 3ml | 3ml | 3ml |
| Agua | 200 | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l |
| NAD | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l | 200m*l |
| Pat.1 | - | - | - | - | - | - | - | - |
| Pat.2 | - | 5m*l | - | - | - | - | - | - |
| Pat.3 | - | - | 5m*l | - | - | - | - | - |
| Pat.4 | - | - | - | 5m*l | - | - | - | - |
| Pat.5 | - | - | - | - | 5m*l | - | - | - |
| Pat.6 | - | - | - | - | - | 5m*l | - | - |
| M 1/20 | - | - | - | - | - | - | 5m*l | - |
| M 1/40 | - | - | - | - | - | - | - | 5m*l |
| Añadimos 2 m*l de enzima alcohol deshidrogenasa | ||||||||
| Agitamos suavemente | ||||||||
| Incubar a 25ºC durante 70 min | ||||||||
| Medimos la absorbancia a 340nm | ||||||||
La tabla correspondiente a las absorbancias de los patrones y muestras son:
| Abs 340nm | Abs - blanco (Y) | Concentracion | Conc Tubo | |
| 1 | -0.107 | |||
| 2 | 0.554 | 0.661 | 100 | 0.3405 |
| 3 | 0.509 | 0.616 | 50 | 0.1702 |
| 4 | -0.046 | 0.061 | 25 | 0.0851 |
| 5 | -0.073 | 0.034 | 12 | 0.0425 |
| 6 | -0.048 | 0.059 | 6 | 0.0212 |
| 7 | 0.725 | 0.832 | 1/20 | |
| 8 | 0.513 | 0.620 | 1/40 |
La recta de ajuste es: Y = a+ bX
Donde Y ea la absorbancia y X es la Concentración. Como sabemos las absorbancias de las muestras, calculamos las concentraciones.
Para la muestra 1 tenemos:
Para la muestra 2 tenemos:
Este es el procedimiento a realizar para el calculo de etanol en la muestra de vino. Siendo el V1 y V2 el % en volumen.
PRÁCTICA 5:
AMILASA DE HARINA DE TRIGO: EXTRACCIÓN, VALORACIÓN Y ESTUDIO CINÉTICO.
OBJETIVO:
Determinar las condiciones óptimas y los parámetros cinéticos de un enzima.
Para valorar la actividad enzimática se utiliza el ácido 3,5-dinitrosalicílico que se reduce a ácido nitroaminosalicílico en presencia de maltosa, y adquiere un color rojo. Mediante la intensidad del color se puede calcular la velocidad de reacción.
Esta práctica se divide en dos partes:
1.- Determinación de la concentación de proteínas en los extractos.
Se pesan 2 g. de harina y después de añadir 20 ml. de tampón se realiza la estracción a temperatura ambiente.Se centrifuga a 3000 rpm durante 5 min. y se recoge el sobrenadante.
La concentración del enzima en el extracto será proporcional a la concentración de proteínas totales las cuales se usan como base de referencia al hacer diluciones del extracto.
Las proteínas se determinan por el método de Biuret.
| TUBO | P2 | P4 | P8 | H1 | H2 | B |
| BSA(10 mg/ml) | 0.2 | 0.4 | 0.8 | - | - | - |
| Extracto de harina | - | - | - | 1 | 2 | - |
| Agua destilada | 1.8 | 1.6 | 1.2 | 1 | - | 2 |
| Reactivo de Biuret | 4 | 4 | 4 | 4 | 4 | 4 |
Se mide con el fotocolorímetro la absorbancia de cada tubo y lo representamos
frente a su contenido en proteínas. Por interpolación calculamos el contenido de proteínas de los tubos H1 y H2.
| Mg BSA | Absorbancia (540 nm) | Absorbancia (540 nm) | |
| PATRÓN 1 | 1.2 | 0.117 | 0.092 |
| PATRÓN 2 | 2.4 | 0.222 | 0.197 |
| PATRÓN 3 | 4.8 | 0.39 | 0.365 |
| MUESTRA 1 | 1.2 | 0.128 | 0.103 |
| MUESTRA 2 | 1.7 | 0.186 | 0.161 |
| BLANCO | 0.025 |
La ecuación de la recta patrón es:
Y = 0.075 X - 0.017 r = 0.998
El resultado medio es: 1.45 mg/ml.
2.- Estudio de la velocidad de la reacción en función de la concentración de sustrato. Determinar la velocidad máxima.
Es necesario realizar la práctica siguiendo la siguiente tabla:
-
Ensayo de actividad.
| 1 | 2 | 3 | 4 | 5 | B1 | B2 | B3 | B4 |
| Enviado por: | Sergio |
| Idioma: | castellano |
| País: | España |