Biología
Virología
VIROLOGÍA - prof Filip Lim
Tema 21: Características generales de los Virus:
-
Historia:
-
La viruela es el agente virico mas antigüo conocido. Desde el Siglo X hasta 1979, siendo la unica enfermedad humana erradicada.
-
Vaccina como Vacuna: en 1718 Lady Montagu fue la primera en usar una “vacuna”, usando pus de algunos animales o humanos infectados en sus propios hijos. Fue a finales de ese siglo en el que Jenner lo hizo definitvamente.
-
En 1892 se distinguen entre enfermedades bacterianas y “otras” por agentes que no quedaban atrapados en el filtro y por tanto eran mas pequeños.
-
Los postulados de Koch se aplicaron en virologia, pero no se cumplen como en bacterias (ejemplo: papiloma virus y cancer).
-
El acido nucleico se determina como portador de la información.
-
Infectan cualquier tipo de celula, eukarya, bacteria o archaea.
-
Propiedades generales:
-
Un virus es un biosistema elemental que:
-
posee genoma (ácido nucleico).
-
capacidad de adaptación a cambios medioa
mbientales. -
no puede capturar y almacenar energía libre.
-
no es funcionalmente activo fuera de su célula huésped.
-
No son tangibles. Una especie viral y un virus no son lo mismo. Hay una gran variabilidad genetica en los viriones.
-
Según ICTV (International Committee on Taxonomy of Viruses) NO SON SERES VIVOS, pues toman prestadas caracteristicas funcionales de una celula mayor o mas compleja.
-
Las categorias taxonomicas son:
-
especie
-
género
-
familia
-
orden
-
no hay mas categorias porque al ser tan simples no se pueden agrupar en grupos mayores en funcion a caracteristicas comunes.
-
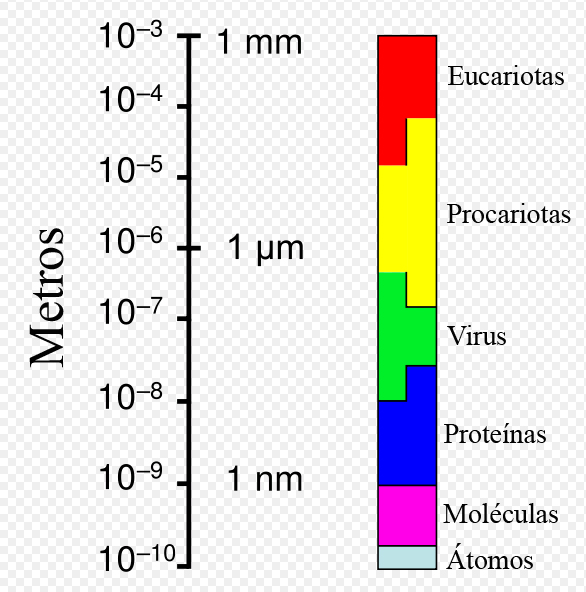
La estructura es, de mayor a menor:
-
Acido nucleico y enzimas (si las hay)
-
Capside (capsula proteica)
-
Tegumento
-
Envoltura lipidica (sin ella no hay tegumento).
-
Hay 3 tipos de estructuras viricas:
-
Helicoidal (alargada)
-
Icosaedrica (forma de icosaedro)
-
Complejas (estilo fago)
-
Tamaño y forma muy variables.
-
Hay virus muy pequeños, y los hay de mayor tamaño que algunas bacterias.
-
Genomas viricos
-
Tamaño del genoma: desde 3,2kb (Parvovirus) hasta 1,2Mb (Mimivirus).
-
El genoma puede ser RNA o DNA.
-
Sus genomas pueden ser:
-
Cadenas simples
-
Doble circular
-
Lineal segmentado/multipartido.
-
Tecnicas de ennumeración de virus:
-
Crecimiento: dependiendo del tipo de virus, la fuente de crecimiento y purificación puede variar: cultivos celulares (animales, plantas, bacterias), tejidos animales o de plantas, embriones…
-
Purificación: el vius puede encontrarse mayoritariamente en el interior de una celula o en el exterior.
-
Cuantificación:
-
Partículas físicas por microscopía electrónica, analisis de proteína, RNA, DNA, prueba de hemaglutinación...
-
Partículas infectivas por detección de transferencia génica (inmunocitoquímica, actividad enzimática).
-
Partículas productivas enumeradas en ensayos de placas

de lisis o titulación por el punto final. -
Términos importantes:
-
UFP: unidades formadoras de placas (pfu en ingles)
-
MDI: multiplicidad de infección (moi en ingles)
-
DL50: dosis letal media para infectar animales (LD50 en ingles)
-
particula / ufp: medida de la infectividad
-
Ciclo infectivo de un virus: procesos comunes durante la infeccion.
-
Fijación (absorción)
-
Penetración.
-
Fase de ECLIPSE / latencia (el virus ha entrado y se ha liberado el material genético. En esta fase el virus no es infectivo para otras celulas)
-
Replicación del genoma.
-
Transcripción y Traducción.
-
Fase de Maduración (Poco a poco van produciéndose y rellenándose las partículas virales).
-
Empaquetamiento del genoma (suele ser simultáneo al ensamblaje).
-
Liberación.
-
Rendimiento: entre varias y varias miles de partículas por célula infectada
-
Varias formas de multiplicación viral:
-
Lisis de la celula huesped (en algunos casos, depende de factores del hospedador: hormonas, anticuerpos.)
-
Persistencia (todas las células producen poco virus ó unas pocas producen mucho y mueren por la infección.)
-
Latencia (desaparece el virus casi por completo y subsiste en forma silenciosa. Factores externos lo hacen reactivarse.)
-
Localización del genoma viral:
-
citoplásmica
-
nuclear - episomal ó integrado (aleatoria ó dirigida)
-
Cuando hablamos de infecciones a animales, consideramos: infecciones agudas, crónicas, recurrentes…
-
APOPTOSIS inducida o inhibida por el virus.
-
Clasificación y Clasificación Molecular Biológica
-
Al principio, según enfermedades provocadas (tejido, modo de infecci
ón, transmisión) -
Características que se consideran para la taxonomía viral:
-
naturaleza del huésped (animal, planta, bacteria)
-
tipo de ácido nucleico (DNA o RNA).
-
número de cadenas.
-
simetría de la cápsida.
-
presencia o no de envuelta.
-
diámetro de la nucleocápsida o del virión.
-
simetría.
-
respuesta inmune que activa.
-
localización intracelular de la replicación viral.
-
tipo de enfermedad que causa o modo de transmisión.
-
Actualmente (2008), la ICTV divide a los virus en 5 órdenes, 82 familias, más de 307 géneros y de 2083 especies según el sistema de Baltimore u otros criterios. “Una especie de virus es una clase politética que constituye un linaje de replicación y que ocupa un nicho ecológico particular”
-
Nombres:
-
Orden: acaba en virales (Caudovirales, Herpesvirales, Mononegavirales, Nidovirales, and Picornavirales)
-
Familia: acaba en viridae
-
subfamilia: virinae
-
el género y especie en virus.
-
Ortografía: el nombre aparece en itálica y la primera letra de la primera palabra en Mayúscula, siempre que nos refiramos al virus como taxón y no como partícula
-
Partículas Subvíricas y Priones
-
VIROIDES - virus de RNA circular pero sin proteína
-
SATELITES - un virus satélite utiliza tanto la célula en la que se reproduce como el virus del que depende para su reproducción. El virus satélite del mosaico del tabaco utiliza al virus del mosaico del tabaco y empeora la enfermedad en la planta
-
Virusoides - RNA circular como los viroides pero dependen de otro virus
-
Virofagos - Sputnik inhibe la replicación del mamavirus
-
PRION (Proteinaceous infectious particle). El agente infeccioso es una proteína de 33-35 kDa. Al parecer actúa produciendo un cambio conformacional de una proteína precursora celular PrP. Además de la encefalopatía espongiforme bovina, se encuentra el SCRAPIE en ovejas o el KURU o Síndrome de CREUTZFELDT-JAKOB en humanos.
-
Teorías sobre el origen y evolución de los virus
-
Los virus infectan los 3 dominios de Vida: Archaea, Bacteria y Eucariota, indicando un origen muy temprano en la evolución de vida. Sin embargo, los virus actuales contienen proteínas, y por lo tanto deberían haber aparecido después de la ribosoma (después de la aparición de células).Las diferencias existentes entre los diferentes tipos víricos indican más de una forma posible en el origen de los virus. Las caracteristicas comunes de muchas de las especies víricas actuales pueden provenir de especies comunes anteriores
-
Evolución por Escape: los virus provienen del material genético celular que, de alguna forma, lograra escapar del control celular para poder perpetuarse de forma independiente. Esta teoría va apoyada en el hecho de que algunos virus, como retrovirus, tienen secuencias muy similares a genes de organismos superiores
-
Evolución Retrógrada: algún virus complejo (vaccinia, herpes) pudo ser originariamente una célula o proto-célula de RNA que parasitara otra de mayor complejidad y que, poco a poco, se fuera haciendo más específica, perdiendo estructuras para garantizar su efectividad en el crecimiento. Los virus de RNA, al ser parasitos de las células ganadoras de evolución, podrían ser los unicos restos de células de RNA que se eliminaron en el concurso selectivo. Los mimivirus podrían ser los restos de células de DNA de un cuarto dominio extinguido.
TEMA 22: Virus Bacterianos:
-
Generalidades
-
Fagos: entidades biologicas mas numerosas (hasta 109/ml en algunas zonas marinas, el 70% de las bacterias marinas infectadas).
-
Los bacteriofagos podrían mantener el equilibrio de las bacterias en el ecosistema.
-
Fagoterapia, agua con propiedades curativas
-
Clasificación
-
Bacteriófagos con genoma de cadena sencilla
-
Fagos con RNA
-
Fagos muy sencillos con 3-4 genes, pueden infectar Enterobacterias.
-
RNA que actúa como mensajero.
-
Se sintetiza inmediatamente, tras la infección, la RNA replicasa.
-
Se sintetiza formas intermedias +RNA (FORMA REPLICATIVA):
-
de la forma -RNA se sintetiza nuevamente miles de copias +RNA.
-
unas copias +RNA actúan como mensajeros y otras vuelven al inicio del ciclo.
-
Ejemplo: MS2 (infecta E.coli, primero genoma secuenciado, primero gen secuenciado (CUBIERTA))
-
Una proteína (MADURACIÓN) colabora en la adsorción, y otros 3 genes codifican para la RNA replicasa, proteína de la CUBIERTA y la proteína de LISIS.
-
La proteína LISIS está codificada por un gen que solapa con el de la cubierta y el de la replicasa (frecuente en virus pequeños).
-
Sólo cuando se ha sintetizado la proteína de la cubierta, el RNA se “relaja” y se hace accesible el sitio de inicio de la proteína de lisis (se evita la muerte prematura de la célula).
-
Fagos con DNA monocatenario
-
M13 infecta Enterobacterias
-
Simetría helicoidal. Tiene 6 nm de diámetro y 860 de longitud.
-
Infectan a través de La Pili.
-
No es lítico, infección de forma permanente que produce fagos continuamente
-
Posee DNA circular (+), se forman ciertos bucles por apareamientos de bases complementarias
-
El ciclo replicativo es complejo: mecanismo de CIRCULO RODANTE.
-
En algunos casos puede integrarse en el genoma bacteriano.
-
Solapamiento de genes
-
FORMAS REPLICATIVAS de dsDNA. El DNA (-) producido puede utilizarse para sintetizar mRNA. Además de virones normales, todas las preparaciones contienen viriones con DNA de: menos de un genoma; varios genomas; genomas defectivos
-
BIOTECNOLOGÍA: secuenciación de DNA, vector de expresión, “display” de proteinas → “nanowires”
-
FX174 infecta Enterobacterias
-
Simetría icosaédrica con proyecciones en algunos vértices.
-
Tiene ssDNA (+).
-
Primer genoma secuenciado totalmente por FREDERICK SANGER en 1977.
-
También tiene genes solapantes.
-
La replicación se realiza también mediante CIRCULO RODANTE.
-
En el proceso de replicación del círculo rodante, la proteína A produce un nick en el extremo 3' de la cadena (+), convirtiéndose en cebador (primer) para iniciar la síntesis de nuevas cadenas lineales de ssDNA, mediante rotación de la estructura.
-
Al completar una vuelta, la proteína A vuelve a cortar y ligar los extremos.
-
Fagos con DNA de cadena doble
-
Muchos son del orden Caudovirales:
-
Virión sin envoltura
-
Cápsida icosaédrica (CABEZA) proteica que contiene una molécula de dsDNA.
-
Un tubo helicoidal para la adsorción e inyección del DNA dentro de la célula huesped (COLA).
-
Un CONECTOR que une ambos elementos.
-
LAMBDA (l)
-
Genoma DNA lineal bicatenario, con extremos cohesivos (COS): en posición 5' de cada extremo tiene unos 12 nucleótidos sin aparear
-
Adsorbción mediante receptores codificados por el gen lamB de E. coli. Sirven normalmente para la entrada de maltosa y se expresan sólamente en presencia de maltosa en el medio.
-
Interacción de lambda con los receptores se mejora con iones de magnesio Mg++
-
Al infectarse una bacteria, el DNA del fago se circulariza y empieza la expresión del genoma.
-
Replicación lítica primero en forma q (o Cairns), y luego círculo rodante
-
LISOGENIA
-
Fago ATEMPERADO, puede entrar en ciclo LISOGÉNICO, el DNA viral esta integrado en el genoma celular y se replica con el celular. En este estado el virus se denomina como PROFAGO. La célula se hace INMUNE contra otra infección por el mismo tipo de fago.
-
La integración del fago (sitio att) ocurre en un sitio único del cromosoma bacteriano (distinto a los extremos COS).
-
mientras dura la lisogenia, procedimientos que dañen el DNA, como la luz UV, pueden inactivar el profago y la célula será CURADA completamente.
-
En el genoma viral, el producto de cI (REPRESOR LAMBDA) inhibe la transcripción del gen cro, (y del resto de las proteínas virales).
-
En el genoma viral, hay 3 sitios (OR1, OR2, OR3) donde el Represor puede unirse: primero se une al sitio 1, esto favorece la unión al segundo Represor al sitio 2 (porque el Represor puede formar dimeros) y, finalmente, al sitio 3 cuando hay altas concentraciones de Represor. El sitio 3 bloquea la transcripción del Represor (gen cI) y asi autorregula el nivel de Represor. De forma completamente inversa se une la proteina Cro a esos sitios. La preferencia de la proteina Cro para unirse al sitio 3 bloquea la transcripción del Represor (gen cI) y elimina la posibilidad de lisogenia.
-
La inducción del ciclo lítico se realiza mediante agentes que dañen el DNA bacteriano, como la luz UV. Se activa el mecanismo de reparación de la bacteria denominado SOS. Una de las proteínas implicadas, la RecA actúa como proteasa degradando a la proteina Represor, permitiendo la expresión de Cro y los otros genes del ciclo lítico.
-
(2) ENTEROBACTERIOFAGO T4:
-
La serie T: se enfocaron los estudios en 7 fagos líticos (T1 - T7) para poder comparar resultados:
-
Autónomamente virulentos ( T-pares y T5) - provocan cese del metabolismo bacteriano durante la infección
-
Virulento dependientes (T1, T3 y T7) - dependen de la continuación del metabolismo bacteriano durante el ciclo lítico
-
Fagos T-pares: el genoma no contiene citosina sino 5'- hidroximetilcitosina, que evita la degradación por nucleasas bacterianas
-
(2) T4:
-
Las moléculas replicadas se aparean en los extremos y formando una molécula de DNA más larga denominada concatámero.
-
Concatémeros de T4 se cortan por una enzima que no corta sitios específicos, sino que lo hace cuando la cabeza del virión está llena. (en cambio la enzima del T7 reconoce sitios específicos sobre el DNA)
-
Los genomas lineales de T4 existen en permutaciones circulares
-
(3) T7:
-
Replicación y regulación similar a T4, pero bastante más pequeño y simple.
-
Codifica sus propias DNA y RNA polimerasas (usos en biotecnología)
-
Solapamientos genéticos
-
Mediante repeticiones terminales se forman concatémeros similares a los deT4, pero el corte posterior de los concatémeros es específico.
-
(4) FAGO P1:
-
Como T4, tiene permutaciónes circulares.
-
Como lambda, puede entrar en lisogenia tras circularizarse, pero NO SE INTEGRA en el genoma bacteriano (infecta E.coli)
-
Debido al origin de replicación viral, en el estado lisogénico, la forma circular del genoma (codifica para un represor) se replica junto a la bacteria
-
Está implicado en transducción: Puede encapsidar hasta un 2-2,5% del genoma de la bacteria. El origin de replicación de P1 se ha empleado para clonar y fragmentos grandes de DNA (hasta 300 Kb), en vectores llamados PACs (P1 artificial chromosomes) en la secuenciación del genoma humano.
-
(5) ENTEROBACTERIOFAGO: Mu:
-
El nombre de Mu se debe a que es un fago MUTADOR
-
Tras la infección el DNA circulariza y se puede producir ciclo lítico ó lisogénico (ambos requieren integración del genoma).
-
Para la integración se corta de forma “cohesiva” el DNA celular, con 5 bases desplazadas, que, al integrarse el fago, se rellenarán, duplicándose en 5 bases el DNA celular.
-
Actúa como un elemento TRANSPOSÓNICO.
-
El DNA viral integrado se corta llevando DNA celular para llenar la cápsida
-
Generalidades
-
Multiplicación: adsorción, penetración, descapsidación, replicación, transcripción, traducción, ensamblaje de las cápsidas y liberación de virus.
-
Liberación por lisis (influenza) o gemación (HIV)
-
Latencia (HSV)
-
Interacción con el sistema inmune
-
Algunos virus están estrechamente relacionados con procesos oncogénicos
-
La mayoría de los virus estudiados pueden modificar la muerte celular programada (apoptosis)
-
Detección:
-
Metodos Directos:
-
Inmunodeteccion:
-
Directa o indirecta (con antibodies marcados. La segunda usa anticuerpos secundarios (ventaja exclusivamente economica)).
-
ELISA (Enzyme-Linked ImmunoSorbent Assay): consiste en usar una placa de plastico con pozillos en los que hay un sustrato con anticuerpos afines al virus)
-
Otros: PCR, Microscopía electronica…
-
Serologicos: No detecta al virus, sino los anticuerpos que reaccionan con sus antigenos:
-
Estado de la infección con virus que no pueden ser cultivados (ej: HCV)
-
Estado del sistema inmune frente a la infección.
-
Crecimiento y Purificación:
-
Principalmente 3 métodos para crecer y purificar virus:
-
Cultivos celulares (el más común): en líneas celulares transformadas. Para detección existen celulas reporteras(Ej: gen de la Luciferasa. Al insertarse en el DNA celular el promotor virico actua como promotor de otros genes celulares (en este caso, Luciferasa) y se empieza a sintetizar esos genes a tutiplen por la propia accion del virus, lo que siempre es un buen indicador).
-
Inoculación en animales (muy costoso y como último recurso)
-
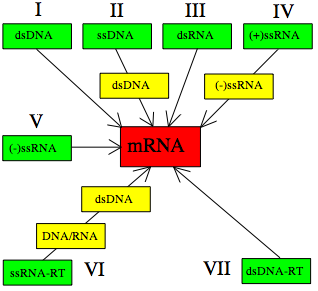
Cultivos sobre embriones (normalmente en huevos, como con el virus de la gripe). Clasificación de Baltimore:
-
Adsorción
-
Muchos virus tienen especificidad por un (o pocos) tipo concreto de células (rhinovirus, poliovirus…)
-
Los receptores celulares para virus suelen agruparse en tipos de moléculas concretos, siendo la SUPERFAMILIA DE LAS INMUNOGLOBULINAS, la más numerosa: CD4 para HIV; ICAM para rhinovirus; HveB para HSV…
-
En muchos casos, bloqueando esta interacción (mediante anticuerpos o fármacos), se inhibe la infección.
-
Otros virus tienen adsorción menos específica y modos de entrar mas generales….
-
Penetración y descapsidación
-
Algunos virus - se elimina la cápsida y se libera el ácido nucleico.
-
El procedimiento final puede variar con el tipo viral (algunas veces depende de pH.)
-
Con virus desnudos se puede producir un cambio conformacional durante la adsorción.
-
En virus con envuelta - fusión con la membrana celular.
-
En algunos casos, se produce una inclusión del virión en la cascada endosomal - escape posterior necesario.
-
Estrategias Replicativas
-
Virus de DNA
-
Muchos virus inhiben la síntesis de macromoléculas celular para quedarse con toda la maquinaria de transcripción y traducción para su ciclo viral. El proceso de inhibición de la síntesis de proteínas celular por el virus se denomina SHUT-OFF.
-
Sin embargo, algunos virus pueden, incluso, estimular la síntesis de DNA celular, llegando, incluso, a favorecer la transformación celular…
-
Replicación no siempre en el núcleo celular - los poxvirus (vaccinia, viruela, genomas de 190 kb)
-
Virus de RNA
-
Estos virus tienen estrategias replicativas más variadas que los virus DNA, según si hablamos de virus de ssRNA (+), (-), ó dsRNA.
-
Retrovirus - TRANSCRIPTASA INVERSA (RT, reverse transcrpitase) que pasa RNA (+) a DNA
-
Replicación de RNA normalmente en citoplasma por una REPLICASA viral
-
Influenza - nucleocápsida va al nucleo donde se replica el RNA
-
RNA polimerasas (transcriptasa) que transcriben un RNA (-) a (+) para que éste pueda ejercer de mRNA.
-
Algunos virus traducen un polipéptido que luego corta con enzimas específicas.
-
Traducción y ensamblaje de las cápsidas virales
-
Virus simples, una fase síntesis de proteínas
-
Virus complejos - distintas fases de síntesis de proteinas
-
IE (Inmediatamente te

mprana) -
E (Temprana)…………………… proteinas procedentes de genes IE, E están implicadas en la replicación viral y su regulación
-
L (Tardía)………………………… componentes estructurales del virus
-
Normalmente, el ensamblaje se produce de manera espontánea al sintetizarse las proteínas estructurales.
-
Tras sintetizarse la PROCÁPSIDA (cápsida vacía), lo siguiente que suele ocurrir es que ésta se llena con el genoma viral.
-
LA MORFOGÉNESIS (formación de nuevas partículas virales) varía: muchos virus RNA en citoplasma, en factorías específicas, muchas veces asociadas a membranas (ej: poliovirus); virus DNA -HSV en el núcleo, poxvirus en el citoplasma.
-
Liberación de los viriones
-
Por término general, los virus desnudos suelen lisar la célula al producir la progenie, mientras que los virus con envuelta pueden liberarse durante cierto tiempo manteniendo a la célula viable.
-
En algunos casos donde el virus con envuelta posee glicoproteínas virales, éstas se pueden unir a membranas celulares y cuando el virus “roba” esta membrana, se lleva en ella dichas proteínas. Estas membranas celulares NO tienen por qué ser la citoplasmática (por ejemplo reticulo endoplasmático).
-
Poxvirus (ej: viruela)
-
Viruela es un miembro de la familia Poxviridae
-
Vaccinia es otro miembro, se usó como vacuna de viruela: virus ATENUADO (esto es, virus al que se la inhabilita la capacidad de virulencia. Se usa Vaccinia porque poose antigenos, pero no capacidad de enfermar al huesped)
-
dsDNA lineal con envuelta (grupo I)
-
Viriones muy grandes - 300 nm, forma de ladrillo, virus animales mas grandes.
-
Replicación en citoplasma - rápida (12 horas).
-
Movilidad dentro la célula utilizando “cohetes de actina”.
-
Herpesvirus
-
8 miembros de la familia de Herpesviridae conocidos en humano:
-
HSV-1, HSV-2, Herpes simplex virus tipo 1 (labial) y tipo 2 (genital), tal vez > 70% personas tienen HSV-1
-
VZV Varicella Zoster: primera infección “varicela” (normalmente infancia), reactivación “herpes zoster”
-
EBV Epstein Barr Virus: virus del beso, está en casi todas las personas sin causar ninguna enfermedad, pero puede provocar mononucleosis infecciosa, linfoma de Burkitt... Puede transformar linfocitos fácilmente en el laboratorio
-
CMV CitoMegaloVirus: infección productiva genera células gigantes, 50-80% personas tiene CMV, la mayoria sin sintomas
-
HHV-6, 7, 8: recien descubiertos, linfotróficos. HHV-8 responsable para Sarcoma de Kaposi
-
Subfamilias:
-
ALFA herpesvirus: neurotrópicos (HSV-1, HSV-2, VZV), replicación rápida.
-
BETA: leucocitos, replicación mas lenta (CMV, HHV-6 y 7).
-
GAMMA: leucocitos, replicación velocidad variable (EBV, HHV-8).
-
Genomas grandes (HSV-1 tamaño 150 kb, ~ 80 genes)
-
3 clases de genes:
-
IE (immediate-early)
-
E (early)
-
L (late)
-
Enzimas del metabolismo de ácidos nucleicos - timidina quinasa, que sintetiza los nucleotidos del herpesvirus.
-
Aciclovir actúa como “prodroga”. El aciclovir por si solo no es toxico, pero en presencia de timidina quinasa SI. Asi, destruye todas las celulas infectadas evitando la progresion de la misma. Hay que tomarlo al menor sintoma!
-
Latencia en forma episomal (fuera del genoma de la celula) en neuronas.(alfaherpesvirus en neuronas)
-
iniciación por sistema inmune(¿?), mecanismo molecular desconocido
-
reactivación por estrés, UV, mecanismo molecular desconocido
-
Estructura de huevo frito (yema = capsida).
-
Membrana con tegumento, glicoproteinas.
-
Tegumento: contiene proteinas multifuncionales. En tipo 1:
-
VP16: factor de transcripcion, necesario para expresar genes IE.
-
vhs: degrada mRNA de la celula.
-
VP22: transporte?
-
Cápsida de proteina, sin envoltura, virion ~100 nm
-
Genoma - dsDNA, lineal ~35 kb
-
No integra en el genoma celular
-
Replica como elemento episomal en el nucleo
-
Cápsida de proteina, sin envoltura, virion ~20 nm
-
No replicativo - parásito de AdV o HSV
-
Genoma pequeño - ssDNA lineal, ambas formas(+) y (-) se encuentran en viriones
-
Integración específica en el genoma celular (Crom 19)
-
Persistencia episomal en células quiescentes
-
No patogénico, poca respuesta inmune
-
Replica en keratinocitos, provoca verrugas.
-
Genoma dsDNA circular de 8 kb, genes E y L
-
Expresión de genes L1, L2 solamente en keratinocitos durante diferenciación (capas superficiales) - evitar sistema inmune
-
Replicación lenta, cultivo difícil
-
Cancer cervical - HPV16 (+ otros), genes E6 y E7 reprimen supresores de tumores (p53 y Rb)
-
Contaminante en vacuna de polio
-
Genoma dsDNA circular de 5 kb
-
Antigeno T - multifuncional: transcripción, replicación, reprime supresores de tumores (p53 y Rb)
-
Entrada por la nariz, ojos.
-
Solo adenovirus tiene DNA.
-
Solo se conoce el 50% de los virus causantes de la enfermedad.
-
Inactivación con detergentes (lavarse las manos).
-
Mas estables a 30°C que a 37°C
-
Dormir poco → susceptibilidad:
-
RELACIÓN CON EL FRIO - mas resfriados en invierno pero no hay mas frecuencia en paises frios, pocos estudios científicos al respecto. El unico que he localizado se referia a la gripe, y hablaba de una alteracion de aquellos virus RNA con membrana, en la que la membrana se hacia mas rigida con el frio, lo que permitia una mayor supervivencia del virus fuera del huesped.
-
superficie de viriones de proteinas VP1-3
-
interaccionan con un receptor celular ICAM-1 (InterCellular Adhesion Molecule 1)que provoca un cambio conformacional en el virion, abriendo un poro en la membrana celular por donde pasa el genoma del virus
-
genoma ssRNA(+) de ~8 kb, cola de poli-A, sirve como mRNA también y es infeccioso como RNA desnudo (pues se traduce como un mRNA celular)
-
proteina viral (VPg) al extremo 5' en vez del 5' CAP
-
IRES = Internal Ribosome Entry Site, permite traducción en la ausencia de 5'CAP, de
-
una poliproteina que se procesa por proteasas virales → 3 proteinas precursoras P1, P2 y P3
-
que a continuación se cortan mas para dar proteinas estructurales (P1), replicasa, VPg y otras proteinas que modifican la célula → lisis (P2 y P3)
-
la proteasa 2A también corta a factores celulares que unen la ribosoma al CAP de los mRNA - asi elimina la traducción celular (CAP-dependiente) pero dejando la maquinaria intacta para traducción viral (CAP-independiente)
-
30% de los catarros - células epiteliales del tracto respiratorio
-
ssRNA(+) lineal ~ 30 kb. Los virus de RNA con mayor genoma.
-
varios RNA que comparten la misma 3' secuencia y una secuencia de 70 bases (Leader) en su 5' extremo (¿salto de transcripción?) - caracteristica de los virus del orden Nidovirales.
-
SARS 2003
-
Sudden Acute Respiratory Syndrome (Síndrome Respiratorio Agudo Severo).
-
China, Hong Kong, Taiwan, Singapur, Vietnam, EEUU, Canadá (solo 1 caso en España).
-
fuente: Guangdong China, carne de animales silvestres.
-
Sarampion (Morbillivirus), Paperas (Rubulavirus).
-
Serotipo único - vacuna triple (con Rubéola) contiene formas activas pero atenuadas de los virus.
-
Con envuelta, nucleocápsida helicoide/filamentosa pero virion esférico.
-
Genoma ssRNA(-) ~15 kb → 7-8 proteinas.
-
Replicación citoplásmica.
-
afecta mamiferos y aves
-
transmisión por líquido (aerosoles)
-
estable: 1 semana temp corporal, 30 dias a 30°C
-
inactivación por detergentes, desinfectantes (lavandose las manos etc).
-
Orthomyxoviridae: Influenzavirus A, B y C, Isavirus, Thogotovirus
-
Clasificacion
-
Influenza A - serotipos H (hemaglutanina) N (neuraminidasa) (como H1N1, causante de la actual pandemia de gripe porcina)
-
Influenza's B y C
-
Isavirus
-
Thogotovirus.
-
Virion filamentoso 900 nm
-
ssRNA lineal 19 kb
-
Codifica 7 proteinas
-
Replicación citoplásmica
-
RNA replicasa (L)
-
mRNAs con Cap y poli-A
-
Adsorción mediante GP→ endocitosis
-
Lentivirus (VIH)
-


Spumavirus (Simian Foamy Virus) -
Oncovirus
-
alpha (Rous sarcoma virus)
-
beta
-
gamma
-
delta (HTLV Human T-lymphotrophic virus)
-
epsilon
-
Virion~100nm con envuelta
-
TRANSCRIPTASA INVERSA (RTasa)
-
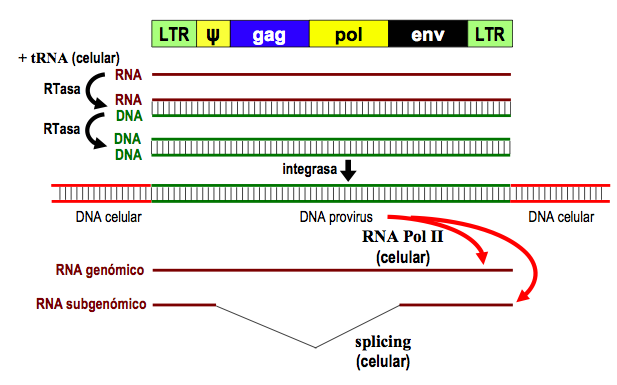
7-15 kb ssRNA(+). Poosen dos copias de este genoma (que no segmentado ni de doble cadena).
-
5' Cap y poli-A
-
minimo de 3 genes:
-
gag (group-specific antigen) proteinas estructurales
-
pol (polimerasa, otras enzimas)
-
env (proteinas de envuelta)
-
Poosen Retrotrascriptasa (trascriptasa inversa/ reversa…) o RTasa.
-
Liberación por gemación, no hay lisis
-
Interferencia viral -resistencia a superinfección, proteinas env ocupan receptores celulares
-
Son los virus de la hepatitis, y son tumorales
-
Hepatitis B-DNA con envuelta, con RTasa
-
pequeños viriones esféricos 40 nm
-
particulas subviricas -filamentos y esferas
-
Infecta higado principalmente, tumores frecuente
-
Genoma:
-
DNA circular, parcialmente doble cadena
-
3,2 kb muy compacto (y muy pequeño)
-
Seguramente son “fosiles” de algun intermediario entre DNA y RNA.
-
Se llaman asi porque en cultivos celulares producen vacuolas enormes en las celulas (espuma).
-
Muchos contienen dsDNA.
-
Existen HIV-1 y HIV-2. Infectan Linfocitos T.
-
glicoproteína gp120 anclada con proteína transmembrana gp41 -interaccionan con CD4 y CXCR4 en la membrana de linfocitos T
-
Se define SIDA cuando < 200 células CD4+/ml.
-
Mata a las celulas tanto por gemacion multiple como al hacer que los Linfocitos citotoxicos maten a la celula infectada o incluso una no infectada pero que tenga antigenos.
-
Linfocitos T tienen un papel crucial en la respuesta inmune, por eso es un virus tan peligroso.
-
Muerte causada por Infecciones oportunistas: Candidiasis, Tuberculosis, Linfoma de Burkitt(EBV), Sarcoma de Kaposi (HHV-8).
-
Tratamiento: Se usan antivirales, que son inhibidors de la actividad integrasa. RTasa o proteasa.La alta tasa de error de la RNA Pol II hace que una vacuna sea muy ineficaz.
| Acidos Nucleicos | Morfologia | Familia | |
| DNA lineal doble cadena (ds) | sin envoltura | colas contractiles | Myoviridae |
| colas no contractiles | Siphoviridae | ||
| Podoviridae | |||
| icosaedricos sin cola | Tectiviridae | ||
| baciliforme | Rudiviridae | ||
| con envoltura | baciliforme | Lipothrixviridae | |
| DNA circular doble cadena | sin envoltura | icosaedricos | Corticoviridae |
| forma de limon | Fuselloviridae | ||
| con envoltura | pleomórficos | Plasmaviridae | |
| DNA circular cadena simple (ss) | sin envoltura | filamentosos | Inoviridae |
| icosaedricos | Microviridae | ||
| RNA lineal ss | sin envoltura | icosaedricos | Leviviridae |
| RNA segmentado ds | con envoltura | esfericos | Cystoviridae |
TEMA 23: Virus Animales I.
Ciclo Replicativo
TEMA 24: Virus Animales II:
Virus con DNA y envoltura membranosa
Virus con DNA sin envoltura membranosa
Adenovirus (familia Adenoviridae)
AAV Virus Asociado a Adenovirus (familia Parvoviridae)
HPV Papilomavirus (familia Papillomaviridae)
SV40 Simian Vacuolating virus 40 (familia Polyomaviri
dae)
Virus con RNA:
Virus con RNA sin envoltura membranosa
Rhinovirus (familia Picornaviridae)
Coronavirus (familia Coronaviridae)
Virus con RNA con envoltura membranosa
Parainfluenzavirus (familia Paramyxoviridae)
Gripe (familia Orthomyxoviridae)
Antigenic Drift (variaciones menores)
virus de RNA tasas de mutación, 10000 veces mayor que el DNA
cuasiespecies
Antigenic Shift (variaciones mayores)
virus influenza A sufren reordenamientos frecuentes
infecciones dobles con virus humano y no humano generan nuevas cepas
Ebola (familia filoviridae (solo 2 generos en esta familia))
TEMA 25: RETROVIRUS
Hay 7 clases:
Caracteristicas generales:
La RTasa funciona de esta manera: Primero sintetiza a partir del RNA una copia complementaria de DNA (usando un tRNA celular a modo de cebador). Entonces empieza a “digerir” o eliminar la hebra de RNA y mientras lo hace, va colocando nuevos nucleotidos de DNA.
Es decir, RTasa tiene 3 funciones: RNA polimerasa inversa (RNA --> DNA), Ribonucleasa y DNA polimerasa.
Una vez hecho esto, ese dsDNA va al nucleo. La Integrasa insertará el DNA en el del huesped, en un sitio especifico o no (depende).
Una vez ocurre eso, la has cagao. El gen puede permanecer silencioso, expresarse o generar un tumor al interferir con la lectura de un gen supresor de tumores (al haberse insertado en medio del gen)...
Si el gen viral se expresa, la Pol II celular sintetiza un mRNA que puede o no sufrir splicing. A partir de este RNA se sintetiza un unico polipeptido. Las Proteasas cortan el polipeptido.
Se hace mediante una estructura secundaria de este RNA que hace que el ribosoma ignore la señal de terminacion (en virus de leucemia). En VIH, el codon de terminacion de una proteina contiene el primer nucleotido de la siguiente.
Liberación:
Ejemplos:
Hepadnaviridae (Hepa (higado) dna (DNA) virus (virus)): DNA virus con RTasa (grupo VII). Sintetizan envueltas vacias
Spumavirus (¿DNA o RNA?)
Lentivirus (VIH):
Genoma tipico de retrovirus.
Descargar
| Enviado por: | Rommel |
| Idioma: | castellano |
| País: | España |
