Biología
Biología Celular
Biología Celular
Obstetricia y Puericultura 2004
Hoy en día, todos aceptamos que el DNA es la molécula que porta la información hereditaria en los organismos vivos actuales (la excepción la constituyen los virus RNA, en caso de que los consideremos como "organismos vivos").
El "dogma" de la biología molecular se refiere al flujo de información en la célula, ilustrándolo de la siguiente manera (figura 1):
Es interesante la reflexión acerca de lo que entendemos como "molécula con información" o "información en la célula". Información es una palabra que se ha tomado desde el ámbito del lenguaje habitual, y para subrayar las diferencias que tiene en relación al ámbito o dominio de la biología celular y molecular, es que se suele utilizar formando la dupla "información molecular". En un intento por clarificar el concepto de información referido a moléculas, despojándolo de todo viso "animista", es que se desarrollará el tema "Expresión génica, transcripción y traducción" haciendo énfasis en las interacciones específicas entre las moléculas que determinan estos procesos.
Aclaremos que sólo trataremos aquí los mecanismos mediante los cuales la "información de la molécula de DNA" determina la secuencia de aminoácidos en una proteína, es decir, la información que codifica para proteínas. No incluiremos en la discusión, el DNA que es transcrito en rRNA, tRNA y otros RNAs -los cuales no son traducidos-, ni tampoco trataremos la "información" estructural del DNA, como por ejemplo la referida al DNA centromérico.
Transcripción y Traducción
Si hacemos temporalmente una concesión, y aceptamos un símil de idiomas y lenguas, podríamos decir que tanto el DNA como el RNA están "escritos en el lenguaje de los nucleótidos" (figura 2 A), y que las proteínas están "escritas en el lenguaje de los aminoácidos" (figura 2B).
A B
Según la Real Academia Española, transcribir significa "copiar en una parte lo escrito en otra". Si pensamos en lo que ocurre en una célula eucarionte, en la cual el DNA se encuentra en el núcleo y el RNA es transportado desde el núcleo hacia el citoplasma, el símil nos lleva a pensar en lo acertado de la elección del término "Transcripción" para indicar el proceso de síntesis de una secuencia de ribonucleótidos (una molécula de RNA) basado en una secuencia de deoxiribonucleótidos (DNA). Lo "escrito" en el núcleo, aparece "escrito en el mismo idioma" en el citoplasma (figura 3). Es sólo un símil, por ejemplo, DNA y RNA no presentan exactamente los mismos monómeros (deoxiribonucleótidos en DNA y ribonucleótidos en RNA; las bases ATGC en DNA, en cambio AUGC en RNA).
De la misma manera, la RAE define traducir como "expresar en una lengua lo que se ha expresado antes en otra". Nuevamente parece acertada la elección del término "Traducción" para aludir al proceso de síntesis de una cadena de aminoácidos (proteína) basada en una secuencia de nucleótidos (RNA) (figura 3).
Transcripción. El proceso
El proceso de síntesis de RNA en la célula eucarionte ocurre en el núcleo, y participan básicamente: DNA, ribonucleótidos, una enzima denominada RNA polimerasa, y una serie de otras proteínas conocidas como factores de transcripción, algunas de cuyas funciones veremos más adelante. En este proceso, una secuencia de DNA sirve de "molde" para la síntesis de RNA; esto es, se sintetiza una molécula de RNA cuya secuencia de bases es complementaria a la secuencia de bases que presenta la hebra de DNA que actúa como molde.
Inicio
En la especie humana, el genoma nuclear está compuesto por 3 x 109 pares de bases (bp), y sólo alrededor de 30 000 genes Varias preguntas son posibles en relación a este dato, una de ellas es...
¿Cómo se explica que sólo determinados segmentos de DNA sean transcritos?
Parte de la respuesta a esta interrogante puede comenzar a deducirse comparando secuencias de DNA que son transcritas, tal como las que se muestran en la figura 4. Estas secuencias fueron alineadas haciendo coincidir en todas ellas el primer nucleótido que es transcrito (+1), o sea, el primer nucleótido que es "copiado" a RNA. En la figura aparece destacada una región en la cual se aprecia una gran similitud en la secuencia de nucleótidos entre todas las secuencias comparadas. Esta región se encuentra aproximadamente en el nucleótido -35, o sea 35 posiciones río arriba (hacia el extremo 5') del primer nucleótido transcrito.
5' 3'
Una hipótesis frente a la petición de explicación por la similitud observada en la secuencia nucleotídica en esta región es que ésta tiene alguna función, que tiene o es algún tipo de información molecular. Una manera de probar esta hipótesis es determinar si se observan o no consecuencias al modificar dicha secuencia (realizando mutaciones puntuales o deleciones). Efectivamente, si se lleva a cabo un experimento en que se deleta una parte de esta secuencia, se observa una importante disminución en la transcripción de las secuencias que se encuentran río abajo (hacia 3'). Como sugieren los resultados, estas secuencias presenta una función relacionada con la promoción de la transcripción, por esto se las bautizó genéricamente como promotores. Los promotores están presentes tanto en procariontes como en eucariontes, precediendo las regiones de DNA que se transcriben. En eucariontes, se observa diferencias en la secuencia y ubicación de los promotores que promueven la transcripción de los diferentes tipos de RNA. En los promotores de eucariontes presentes en genes que codifican para proteínas, se encuentra una secuencia de Ts y Aes denominada por esto, caja TATA. (el ejemplo analizado en la figura 4). En realidad, la caja TATA es uno de los elementos que se puede encontrar en un promotor, habitualmente hay otras secuencias cercanas a la caja TATA que también son elementos del promotor. En los últimos años, se ha determinado que unos pocos genes transcritos por la RNApol II, no presentan caja TATA; en estos se encuentra la secuencia Inr.
Posteriores investigaciones permitieron determinar que a estas secuencias promotoras de la transcripción se unen proteínas de manera específica. La unión de estas. proteínas es requisito para que el promotor funcione como tal, es decir, promueva la transcripción. De hecho, es la falta reconocimiento y, por consiguiente de unión de estas proteínas a un promotor mutado o con alguna deleción, lo que explica la disminución o falta de transcripción en el experimento mencionado más arriba.
Para ilustrar el proceso de transcripción, permaneceremos haciendo referencia a este proceso en organismos eucariontes particularmente, de genes que codifican para proteínas.
¿qué proteínas esperaría usted encontrar unidas directamente a los promotores?
Dada su función de catálisis en la reacción de síntesis de RNA, era esperable encontrar a la RNA polimerasa unida a las secuencias promotoras. Sin embargo, esta enzima no se une directamente a los promotores. Como se esquematiza en la figura 5,en el proceso de transcripción participan una serie de proteínas o complejos proteicos, a los que se les denominada factores de transcripción. Cada uno de estos factores de transcripción se nombra como TF (por Transcription Factor) seguido de un número romano y, una letra. El número romano (I, II o III), indica de cuál de las tres polimerasas es factor. Es así como, para los factores de la RNA polimerasa II, que transcribe genes que codifican para proteínas, tenemos TF IIA, TFIIB, etc.
Como se muestra en el esquema de la figura 5, a la caja TATA se une directamente una proteína conocida como TBP (TATA Binding Protein - proteína de unión a la caja TATA), que es parte del factor de transcripción TFIID (este se compone al menos de 9 subunidades, TBP y otras subunidades pequeñas conocidas como TAFs). Una vez que TBP, y TFIID se unen al DNA, se unen secuencialmente a este complejo, el resto de los factores de transcripción y la RNA polimerasa II.
Es destacable la función de TBP. Esta proteína determina en qué puntos del DNA se ensambla la maquinaria de transcripción, y esto es posible porque esta molécula presenta una determinada disposición espacial de átomos que son capaces de establecer interacciones químicas con otros presentes en la caja TATA en el DNA (interacciones químicas termodinámicamente favorables, estables, se entiende) La figura 6, a la izquierda, muestra un esquema deTBP interactuando con la caja TATA, y a la derecha, un acercamiento que muestra los puentes de hidrógeno en que se basa dicha interacción.
Es la relación y disposición espacial que presentan los átomos en una molécula, lo que determina las interacciones moleculares que ésta puede establecer; es a esto a lo que nos referimos cuando hablamos de información molecular.
Una vez que se ha formado este gran complejo multiproteínas-Caja TATA, comienza la etapa de elongación, pero esto requiere al menos de dos requisitos: la separación de las dos hebras de DNA, por la función helicasa de uno de los factores de transcripción, y la fosforilación masiva d la cola C' terminal de la RNApol II. Esta fosforilación parece afectar la estabilidad de la unión de la RNA polimerasa al complejo, permitiendo que esta avance en la dirección de la transcripción.
Elongación
En el sitio activo de esta enzima, se ubican los nucleótidos de DNA, comenzando con el +1(el primero en ser transcrito) y los ribonucleótidos complementarios. La acción enzimática de la enzima permite la formación del enlace fosfodiéster entre el C 3' del primer ribonucleótido y el 5' del segundo de ellos, tal como se observa en la figura 7.
Las RNAs polimerasas transcriben el DNA de la hebra que "recorren" en dirección 3'-5', tanto en procariontes como en eucariontes. Los ribonucleótidos complementarios se unen al DNA "cabeza abajo", es decir si en el DNA se encuentra hacia la "izquierda" el extremo 3', el ribonucleótido expondrá en ese sentido (hacia la "izquierda") su región del C 5'; el ribonucleótido siguiente ingresará a la cadena de RNA creciente, en la misma disposición espacial (C 5' hacia la "izquierda"), por lo tanto, observaremos que el RNA se sintetiza desde su extremo 5' al 3', es decir será antiparalelo a la hebra de DNA transcrita (figura 7).
Un segmento de DNA puede ser transcrito por varias RNA polimerasas a la vez, las cuales se habrán unido al promotor en momentos diferentes, de tal manera que las que se unieron al promotor más temprano, irán más adelantadas en el proceso que las que se unieron al promotor más tardíamente. Esto se observa al MET como aparece en la figura 8.
La RNApol va recorriendo el DNA, a una velocidad estimada de 50 nts/segundo en procariontes, y 20 nts/segundo en eucariontes. A medida que esto ocurre, el RNA sintetizado, hasta ese momento estrictamente formado por una secuencia de ribonucleótidos complementarios al DNA que sirvió de molde, comienza a ser transformado. Aparentemente las funciones enzimáticas que participan en estas transformaciones -procesamiento- las llevan a cabo proteínas que se asocian a la RNApol.
Adición de un capuchón (Capping): El primer segmento de RNA que asoma del complejo de transcripción corresponde a la región 5' de la molécula, y es aquí donde se observa la primera modificación. Cuando se han sintetizado cerca de 25 nt, un conjunto de enzimas particulares, y distintas de la RNApol, catalizan la adición de un ribonucleótido de guanina -que luego es modificado por una metilación- al ribonucleótido del extremo 5' de la molécula naciente de RNA del cual se ha removido una molécula de fosfato (figura 9). Este "capuchón" molecular se une a proteínas que cumplen una función tanto en el posterior transporte del mensajero hacia el citoplasma como en la traducción de él, según veremos más adelante.
Remoción de intrones (Splicing): El estudio de los genes eucariontes resultó, en muchos aspectos, más complicado que el de los procariontes. Por ejemplo,1) si se busca la secuencia de DNA que codifica para un RNA mensajero de secuencia conocida no es posible encontrarla, y 2) en el núcleo se pueden encontrar RNAs mensajeros de un tipo dado, que son más largos que los citoplasmáticos del mismo tipo, es decir, en el interior del núcleo aparecen RNAs mensajeros con secuencias que finalmente no llegan al citoplasma y, por esto, no codifican para proteína.
Esto se debe a que los genes están codificados en el DNA interrumpidos por secuencias no codificantes denominadas intrones. Las secuencias codificantes entre intrones se denominan exones. Los intrones son transcritos, pero luego escindidos del RNA en un proceso complejo denominado splicing (corte y empalme); en este proceso de splicing, que ocurre durante la transcripción (co-transcripcional), participan agregados macromoleculares formados por proteínas y RNAs pequeños (ribonucleoproteínas), denominados spliceosoma. El spliceosoma reconoce señales presentes en la secuencia de RNA, se une a ellas, corta en dos puntos el transcrito y luego une el extremo del RNA que se encuentra río arriba del sitio de corte más próximo al 5' con el extremo que se encuentra río abajo del sitio de corte más cercano al extremo 3' (ver figura 10). Nuevamente hemos hablado aquí de "señales" haciendo referencia al concepto de información molecular. En este caso, se trata, aparentemente, de interacciones entre las bases del transcrito con las de los RNAs pequeños que forman parte de las diversas subunidades del spliceosoma.
Término
La etapa de elongación, durante la cual ocurre el capping y splicing, continúa hasta que la maquinaria transcripcional encuentra una señal de término en la cual ésta se detiene y libera tanto el DNA templado como el RNA. En los genes procariontes, las señales de término las podemos identificar con mayor facilidad, puesto que presentan, en general, secuencias de consenso. En eucariontes, sin embargo, resultan más difíciles de determinar, puesto que aparentemente lo que está conservado es la presencia de una secuencia de bases que produce un enlentecimiento en el desplazamiento de la maquinaria de transcripción; la combinación de bases capaz de producir este enlentecimiento es muy variada.
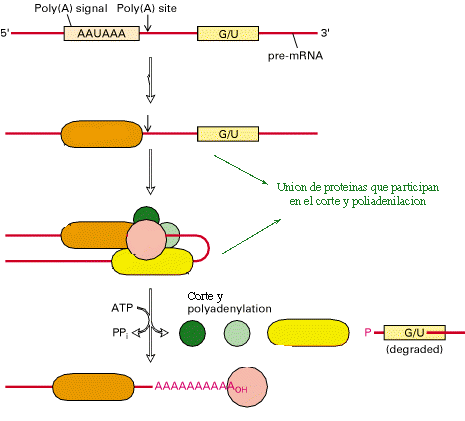
Poliadenilación: Sin embrago, río arriba del término, se encuentra una secuencia ampliamente conservada (AAUAAAA), la cual es reconocida por otro grupo de enzimas que, básicamente, realizan dos modificaciones del transcrito. En primer lugar, lo cortan, y en segundo lugar, al nucleótido que queda en el extremo 3', le agregan una cola de PoliA, es decir una larga secuencia de nucleótidos de adenina (aproximadamente 200 nts) (figura 11). Una de las funciones descritas para la cola de poliA está relacionada con la vida media del mensajero; una disminución de la longitud de la cola, se relaciona con degradación del mensajero.
Esta serie de interacciones, aparentemente, producen una desestabilización de la maquinaria de transcripción, con lo cual, ésta se libera del DNA unos pocos cientos de nucleótidos río abajo del sitio de corte y poliadenilación.
Exportación
Una vez que el mRNA ha sido transcrito y completamente procesado, se encuentra en el núcleo asociado a una serie de proteínas. Entre éstas, algunas portan señales que, permiten su interacción con otras proteínas y con proteínas del complejo de poro nuclear, determinando la apertura de éste y la salida del mRNA maduro hacia el citoplasma (figura 12).
Traducción
Además del RNA mensajero, en el proceso de traducción existen una serie de otras moléculas involucradas. Haremos una breve referencia a un par de ellas.
Los ribosomas: son grandes complejos ribonucleoproteicos (proteínas ribosomales+ rRNA), que se ensamblan en el nucléolo en forma de sub-unidad mayor y sub-unidad menor. La estructura tridimensional de estos agregados macromoleculares es realmente compleja y se ha comenzado a entender mejor luego de la cristalización de estos. Las sub-unidades mayor y menor sólo se unen durante el proceso de traducción durante el cual se encuentran unidos también al mRNA(figura 13),. Cuando se estructura el ribosoma, se constituyen sitios tridimensionales que son verdaderos “bolsillos” moleculares; estos se cuentan en número de tres y se conocen como sitio A, sitio P y sitio E.
Tanto los ribosomas como el proceso de traducción en sí, presentan importantes diferencias entre procariontes y eucariontes. La base del uso de algunos antibióticos como los aminoglicósidos, por ejemplo, se encuentra precisamente en estas diferencias.
En este documento nos limitaremos a ciertos aspectos del proceso de traducción en eucariontes.
El tRNA : es una molécula clave en el proceso de traducción, volviendo al símil del inicio de esta nota, podríamos decir que actúa como traductor, ya que “habla los dos lenguajes, el de los nucleótidos y el de los aminoácidos”. Esto es resultado de la acción de una familia de enzimas llamada aminoacil tRNA sintetasa, las cuales reconocen específicamente la región del anticodón del tRNA (figura14), entre otras, y le transfiere a éste (catalizando la formación de un enlace covalente) en forma epecífica, un aminoácido particular. Se forma así una molécula híbrida RNA-aminoácido conocida como aminoacil-tRNA
Etapa I. Inicio de la traducción
Al llegar el mRNA al citoplasma, el capuchón 5' es reconocido por factores de traducción. Estos factores son un grupo de proteínas, que en conjunto con ribosomas, y aminoacil-tRNAs, entre otros, son indispensables para el proceso de traducción.
En la etapa de inicio de la traducción, al sitio P (o, mejor, al hemisitioP) de la sub-unidad ribosomal menor, se une un aminoacil-tRNA iniciador (que siempre corresponde a un tipo de metionil-tRNA). Este complejo, reconoce y se une al RNA mensajero por el extremo 5', al cual se encuentra unido uno de los factores de inicio de la traducción. Este complejo sub-unidad_ribosomal---metionil-tRNA_iniciador, recorre el mRNA hacia el extremo 3'; el complejo se detiene en el punto en que quedan confrontados la región del anticodón del metionil-tRNA con el triplete AUG (figura 15)
cómo se puede explicar esta detención del complejo? ¿A qué se le llama codón de inicio de la traducción?
La estabilización del complejo está dada por la formación de puentes de hidrógeno entre las bases del anticodón del metionil-tRNA iniciador y el triplete o codón AUG del RNA mensajero. Piense cuál será, entonces, la secuencia presente en e anticodón.
AUG será el codón de inicio de la traducción de todos los mensajeros, puesto que el único aminoacil-tRNA que es capaz de unirse a la subunidad ribosomal menor es el metionil-tRNA de inicio.
Luego de la estabilización del complejo, cuando el codón AUG se encuentra frente al “hemi”sitio P donde se encuentra el metionil-tRNA de inicio, se une la subunidad ribosomal mayor. Ahora se crean en rigor, los sitios A, P y E. En el “piso” del sitio A se expone ahora otro triplete, otro codón, y a él ingresará y será estabilizado, un aminoacil tRNA cuyo anticodón presente una secuencia complementaria. Quedarán entonces muy cercanos los aminoácidos metionina (unido al tRNA iniciador) y el aminoácido unido al tRNA que ingresó al sitio A (figura 16). La sub-unidad ribosomal mayor, que presenta la actividad enzimática peptidiltransferasa, cataliza la formación del enlace peptídico, formando un dipéptido. Éste péptido irá creciendo en la medida en que nuevos aminoacil tRNAs ingresen al sitio A. durante la fase de elongación.
Etapa II. Elongación
Al formarse el enlace peptídico, los aminoácidos quedan unidos al, antes aminoacil, ahora peptidil tRNA que se encuentra en el sitio A. Con esto, se produce un cambio conformacional ene l complejo, se desplaza el ribosoma en relación al mensajero, y el tRNA "descargado" sale del complejo por el sitio E (de Exit, salida); por su parte, el peptidil tRNA qu se encontraba en el sitio A, pasa al sitio P (de Peptidil), y el sitio A queda ahora libre, y exponiendo el siguiente triplete del RNA mensajero.
De la manera que se explicó más arriba, sigue creciendo el polipéptido (figura 17). Es necesario aclarar que en esta etapa de elongación participan una serie de factores a los que no se hará mención aquí.
Si reflexionamos sobre el proceso descrito, nos podemos dar cuenta de que el mRNA se va decodificando de a tres nucleótidos -lo que entendemos por codón-, y que estos tripletes se "leen" de corrido, sin saltarse nucleótidos. Esto último está dado por la conformación de los sitios A y P que "exponen" dos series continuas de tres nucleótidos -tres cada uno, sin nucleótidos entre medio-. Puesto que el aminoácido unido a cada tRNA es el mismo en todos los tRNAs que comparten la misma secuencia en el anticodón, y que son precisamente los nucleótidos del anticodón los que se unen al codón del mensajero, entonces, a cada a codón de éste le corresponde un aminoácido dado. A esta correspondencia entre codones del RNA mensajero y aminoácidos se le denomina código genético (figura 18).
v
Etapa III. Término
La elongación del polipéptido continua hasta que el sitio A es ocupado por uno de tres codones de los denominados codones de término. Cuando esto sucede, en lugar de ingresar al sitio A un aminoacil-tRNA, ingresa una proteína denominada factor de término o liberación el cual produce la hidrólisis de la unión tRNA-péptido, liberandose la proteína, tRNAs, ribosomas y mensajero, con lo cual concluye la síntesis de la proteína. Todo este proceso puede suceder tanto en el citoplasma como asociado a las membranas del RER.
Usualmente, el codón de término no se encuentra al final (extremo3') del RNA mensajero, como tampoco el codón de inicio se encuentra al principio de éste (extremo 5'). Los segmentos del mensajero que se encuentran río arriba del codón de inicio y río abajo del codón de término, se denominan secuencias UTR (no traducidas), y han sido involucradas en diferentes aspectos del control de la expresión génica, tanto en regulación de la vida media del mensajero, como de control a nivel traduccional.
Cabe acotar aquí, que una interesante característica del sistema. Un gen puede ser transcrito simultáneamente por varias RNA polimerasas (figura 8), a su vez, cada RNA mensajero así sintetizado, puede ser traducido simultáneamente por muchos ribosomas, muchas veces (figura 20). Esto determina que en este sistema se pueda sintetizar en un momento dado, una gran cantidad de una proteína codificada sólo por un gen.
Terminar comentando sobre las múltiples etapas en las que se ha observado que existe o puede darse el control de la expresión de los genes, que es uno de los determinantes de que el funcionamiento y capacidad de respuesta de las células y el organismo completo sean compatibles con el estar vivo. Tanto el proceso de transcripción, como el procesamiento del RNA, su salida al citoplasma, vida media y si será o no traducido, y en qué medida, junto a las modificaciones químicas de las proteínas (que regulan su actividad) y el compartimento celular en que se encuentren (que determinan su funcionalidad), son aspectos que se encuentran finamente controlados y coordinados en el proceso de expresión de nuestros genes.
Descargar
| Enviado por: | Seto |
| Idioma: | castellano |
| País: | Chile |
