Biología
Informe sobre el Gen AT5G54160 de Arabidopsis Thaliana
INFORME SOBRE EL GEN AT5G54160 DE ARABIDOPSIS THALIANA
1 - Características de la secuencia del gen en el genoma
Este gen se localiza en el cromosoma 5 del genoma de Arabidopsis Thaliana y codifica para la proteína O-metiltransferasa.
a) Características del gen AT5G54160
Fig 1. Esquema del gen.
Como se puede observar en la figura la orientación del gen en corriente abajo 5' 3' y esta compuesto de cuatro exones y tres intrones. El tamaño del gen, la localización del gen dentro de cromosoma cinco, y las coordenadas del los exones e intrones se muestran el la tabla 1.
| Gen | AT5G54160 |
| Orientación | 5' 3' |
| Tamaño secuencia genómica (PB) | 2368 |
| Tamaño CDS (PB) | 1092 |
| Tamaño cDNA (PB) | 1367 |
| Localización cromosomica | 5 |
| Posición en el cromosoma (NT) | 21999222-2200490 |
| Número de Exones | 4 |
| Coordenadas Exón 1 (NT) | 1-492 |
| Coordenadas Exón 2 (NT) | 1299-1606 |
| Coordenadas Exón 3 (NT) | 1690-1754 |
| Coordenadas Exón 4 (NT) | 1870-2398 |
| Número de Intrones | 3 |
| Coordenadas Intrón 1 (NT) | 493-1298 |
| Coordenadas Intrón 2 (NT) | 1610-1689 |
| Coordenadas Intrón 3 (NT) | 1755-1869 |
| Coordenadas 5' UTR | ene-79 |
| Coordenadas 3' UTR | 2173-2368 |
Tabla 1.
b) Otros datos sobre el gen.
Promotor: debe localizarse cinco prima corriente arriba antes del inicio de la transcripción.
Genes flanqueantes: en imagen dos se puede observar los genes que flanqueantes.

Fig 2. Situación de los genes en el genoma.
2 - Características de la transcripción del gen.
Los distintos cDNAs derivados de los transcritos son los siguientes:

Fig 3. cDNA
Transcritos detectados se han aislado cinco tipos distintos de cDNA para el gen, por lo que se deduce que codifica para cinco mRNA muy parecidos, que se traducen a proteína con funciones similares ya que sólo difieren en la longitud de dos de los cuatro exones.En la tabla dos se especifican el nombre del clon, el tipo de vector y su posición.
| Nombre del clon | Locus | Acceso Gen Bank | Tipo de Vector | Cromosoma | Posición |
| 21406021 | AT5G54160 | AY087297 | 5 | 21999223 pb | |
| 2781393 | AT5G54160 | U70424 | 5 | 21999220 pb | |
| GSLTPGH15ZH04 | AT5G54160 | BX831565 | 5 | 21999242 pb | |
| RAFL06-07-M15 | AT5G54160 | AY062837 | plásmido | 5 | 21999223 pb |
| U17164 | AT5G54160 | AY081565 | plásmido | 5 | 21999301 pb |
Tabla 2.
Característica de la expresión del gen:
a) Expresión en las distintas etapas de desarrollo y en las distintas partes de la planta
-Cuando se miden los niveles absolutos de expresión del gen se observa un nivel nulo o un poco reprimido de en todos los tejidos y órganos de la planta y en los diferentes estadios de desarrollo. Solo se ve un poco de expresión en el primer y segundo internudo, siendo en este ultimo la mayor expresión. En cambio habría que señalar que la represión de la expresión es máxima en la semilla seca.
Expresión en tejidos específicos: hay una pequeña expresión en los órganos reproductores de la flor tanto en los ovarios y las anteras
Expresión tras tratamientos de estrés biótico: a someter a las planta de Arabidopsis a diferentes tratamientos de estrés biótico (Fig 4), se observa expresión del gen estudiado. Tras el estrés provocado por la infección con Elicitors la expresión es máxima según la escala seguida (valor de Ratio 1.86) a las cuatro horas depuse de la infección. En Phitophora Infestans la expresión máxima se da a las doce horas (después de la infección en las hojas de Arabidopsis.
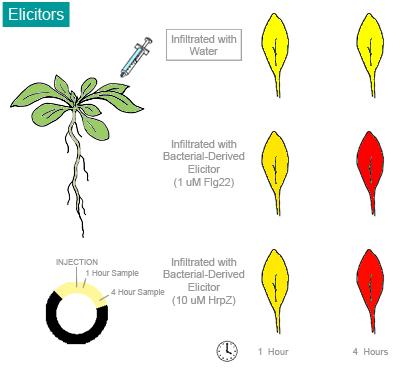
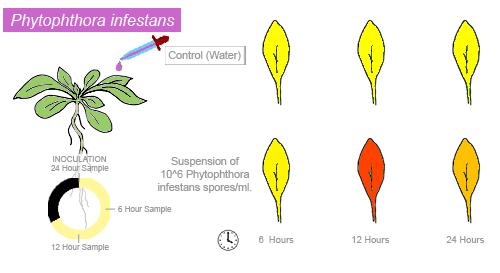
Fig 4. Tratamientos que provocan estrés biótico en Arabidopsis Thaliana.
Expresión tras tratamientos de estrés abiótico: después de someter a las plantas a diferentes tratamiento de estrés algunos de ellos como; frío continuo a 4 ºC en la cámara de frío,, sal (150 mM), manitol (300mM) y la provocación heridas por punción en las hojas, provocó que la expresión del gen fuera máxima (Ration 1.34) a las doce horas del tratamiento en todos los casos anteriormente citados. Si embargo en el transcurso de las doce horas la expresión cayó prácticamente a cero, menos en al tratamiento de frío donde la expresióno fue fuertemente reprimida.
En la figura cinco podemos observar la expresión del gen en los distintos tejidos u órganos, siendo la expresión mas significativa en la raiz.
Fig 5. Expresión del gen en tejidos y órganos de Arabidopsis.
En la siguiente figura podemos ver como cambia la expresión del gen antes distintos tratamientos. El gen puede ser inducido o reprimido según el tratamiento. La manera de cuantificar el cambio de expresión es por el valor del ratio.
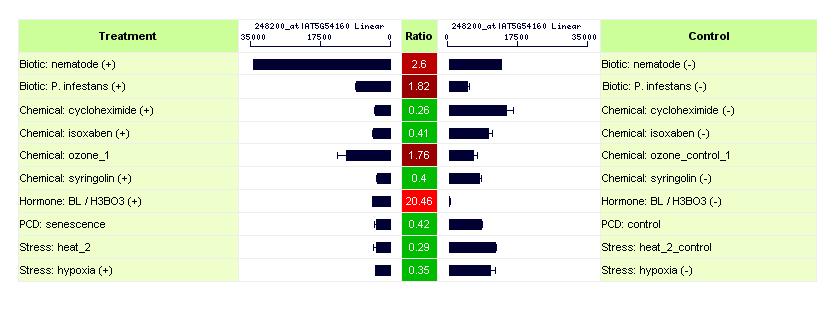
Fig 6. Expresión del gen, tras ser inducido (color rojo), o reprimido (color verde).
4- Características de la traducción del gen.
Longitud de la proteína 363 aa , tiene un peso molecular: 39617, 6.Daltons y su punto isoeléctrico: 5.8107.
Función del gen
El gen AT5G54160 codifica para la O-metiltransferasa, que pertenece a una pequeña familia multigénica en A. thaliana [2]. Entre sus funciones producen la O-metilación de flavonoides, componentes de las plantas esenciales para su desarrollo y supervivencia. En plantas los flavonoides O-metilados son menos reactivos y mas lipofílicos, por lo que aumentan su actividad antimicrobiana y antiviral. Esto se ha podido observar en las figuras cuatro y seis donde la expresión del gen aumenta ante la infección de patógenos. En mamíferos reducen su mutagenicidad y previenen la angiogénesis previniendo la formación de tumores [3].
Otra de las funciones de la O-metiltransferasas está implicada en la biosíntesis de lignina. La lignina es un componente importante de la pared celular de las plantas, especialmente de plantas leñosas donde puede constituir un 20-30% del peso seco [2].
La lignina proporciona a la plantas varias ventajas, como capacidad de transporte de H2O a larga distancia [2], hidrofobicidad a los vasos del xilema y soporte mecánico a los tallos [1]. La lignina deriva principalmente de la polimerización de tres alcoholes hidroxicinnamíticos [1], que dan lugar a las unidades en el grado de metoxidaxión en posiciones 3 y 5 del anillo aromático. Son necesarios dos pasos de metilación en los monolignoles[1]:
-COMT (Ácido Cafeico O-metiltransferasa): produce la O-metilación en el CS del anillo fenólico, dando lugar al precursor de unidades S de lignina, Sinapyl alcohol.
-CCoAOMT 1 (Cafeoil CoA O-metiltransferasa): codificada por el gen AT4G34050 codifica la enzima que actúa conjuntamente con COMT1 para mutilar el C3 del anillo fenólico de los monolignoides, en Arabidopsis.
-CCoAOMT: esta implicada en la biosíntesis de los precursores de lignina G, ya que metila a cafeoil CoA para producir feruloil CoA, el precursor de unidades de lignina G.
Roes et al (2003) clasifican los genes de CCoAOMT en dos clases, siendo CCoAOMT1 el único miembro de la clase 1. Su expresión se restringe a los tejidos vasculares, excepto en plántulas, donde parece que se expresa constitutivamente. En plantas adultas, su expresión es específica en vasos xilemáticos y en fibras interfasciculares, es decir las partes lignificadas de los tallos de Arabidopsis thaliana. Este patrón de expresión es indicativo de su papel en la lignificación [1], unto a COMT1, lo cual no es el caso de otros genes CCoAOMT y COMT, que no se expresan en tallos.
Existen dos isoformas de OMT1, un monoespecífica (principalmente en gimnospermas) y otra biespecífica (principalmente en angiospermas). La OMT1 biespecífica lleva a cabo dos reacciones [2]:
Acido cafeíco Acido telúrico
Acido 5-Hidroxifelúrico Acido Sinápico
En Arabidopsis, OMT1 es biespecífica y está codificada por un solo gen del copia simple. Su patrón de expresión es mayor en tallo, con los niveles intermedios en flores raíz y hojas.
5- Análisis filogenético
. En la figura siete se muestra posibles proteínas homologas de la proteína resultante del gen estudiado. Siguiendo la escala de color que señala la homología respecto a la proteína del gen AT5G54160 (color morado) diría que se trata de una familia génica ya que guarda mucha homología. Otro dato que señala la homología entre las proteínas es la probabilidad N, que en la imagen aparece representado por un número exponencial, tal como 2.5e-90, que cuanto menor es, mayor homología representa esa proteína respecto a la proteína del gen analizado.
Para la búsqueda de genes ortólogos los datos han sido tratados por análisis en la base de datos EMBL-EBI. Aquí hemos comparado la secuencia de aminoácidos de la proteína del gen AT5G54160 con otras proteínas resultante de los demás genes que se muestran en la figura 8 y los resultados de DbClustal, corroboran que hay una alta homología entres ellas, con lo cual apoya el echo de que se trata de una familia génica.
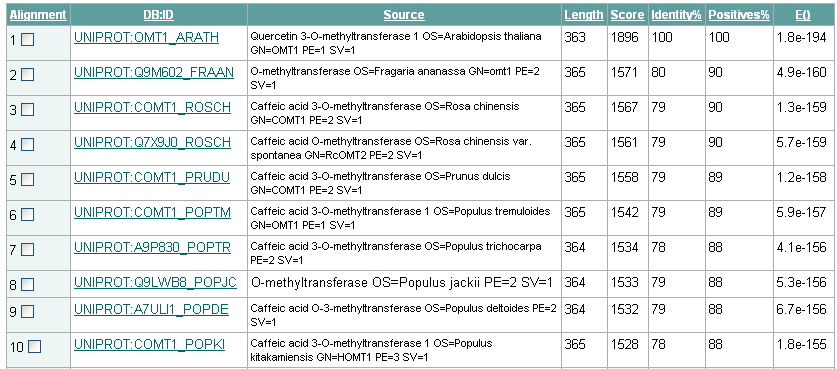
Fig 8.Tabla de homología.
Las proteínas que presentan mayor homología, teniendo mayor probabilidad de ser un gen ortólogo pertenecen a la familia de las rosáceas, como la fresa (Fragaria ananassa), el rosal (rosa chilensis) y el almendro (Prunus dulces).
6- Herramientas para el análisis del gen
De la relación de mutantes nos quedamos con los mutantes SALK que se han insertado en exones, su característica se observan en la tabla 3.
Los mutantes pueden insertarse en cualquier sitio (UTR, promotor, intrón o exón) y pueden afectar a la función de la proteína.
UTR: no hay mutantes que se hayan insertado en el extremo 5'UTR, de haber mutantes el gen se transcribe a RNA pero puede tener problemas en la traducción, si el mutante afecta en el sitio de unión a ribosomas, de manera que no habrá proteína. El extremo 3' UTR es más flexible y hay menos probabilidad de que afecte a la transcripción.
Promotor: de los datos obtenidos ningún mutante se inserto en el promotor por lo que no podemos saber su efecto que podrían ser varios como, aumentar, diminuir la expresión del gen o no hacer nada.
Intrones: los mutantes se pueden insertar en los intrones pero como estos son eliminados en la maduración del RNAm no suelen afectar a la función de la proteína. Aunque pueden afectar al splicing del intrón. También pueden provocar una terminación prematura de la traducción, si el mutante posee una secuencia nucleotídica que al insertarse provoque dicho efecto.
Exones: la manera más probable de que se vea afectada la función de la proteína es que el mutante se inserte en una exón, muy probablemente afectará al dominio funcional de la proteína viéndose su actividad disminuida o completamente eliminada.
| Nombre del mutante | Tipo de mutación | Zona del gen | Nucleótidos | Sentido |
| SALK_135290.45.60.x | inserción | exón | 22000542 | 3'-5' |
| SALK_146526.37.60.x | inserción | exón | 22001539 | 5'-3' |
Tabla 3 Mutantes: relación y descripción física y funcional
BIBLIOGRAFÍA
Artículos consultados.
1 Do, Cao-Trung, Pollet, Brigitte, Thévenin, Johanne, Sibout, Richard, Denoue, Dominique, Barrière, Yves, Lapierre, Catherine, Jouanin, Lise.Both caffeoyl Coenzyme A 3-O-methyltransferase 1 and caffeic acid O-methyltransferase 1 are involved in redundant functions for lignin, flavonoids and sinapoyl malate biosynthesis in Arabidopsis. JournalPlanta 2007 Oct;226(5):1117-29
2 Hong Zhang , Jing Wang, Howard M. Goodman. An Arabidopsis gene encoding a putative 14-3-3-interacting protein,caffeic acidr5-hydroxyferulic acid O-methyltransferase 1. Biochimica et Biophysica Acta 1353_1997.199-202.7 May 1997
3 Ingrid Muzac,* Jing Wang,† Dominique Anzellotti,* Hong Zhang,† and Ragai K. Ibrahim. Functional Expression of an Arabidopsis cDNA Clone Encoding a Flavonol
39-O-Methyltransferase and Characterization of the Gene Product1. rchives of Biochemistry and Biophysics Vol. 375, No. 2, March 15, pp. 385-388, 2000
Descargar
| Enviado por: | Daniel |
| Idioma: | castellano |
| País: | España |
