Biología, Botánica y Zoología
La expresión de la información genética
LA EXPRESIÓN DE LA INFORMACIÓN GENÉTICA: TRANSCRIPCIÓN Y TRADUCCIÓN
1. Concepto de gen.
A. Teoría un gen-un enzima. Tras diversos estudios en la primera mitad del siglo XX, G. Beadle y D. Tatum llegaron a la conclusión de que los genes contienen información para sintetizar proteínas enzimáticas y propusieron la hipótesis de “un gen, un enzima”, lo que se generaliza después como “un gen, una proteína, un carácter”. Dicho de otro modo, los genes (el ADN) se expresan mediante la síntesis de proteínas y éstas catalizan reacciones bioquímicas que son las responsables de los caracteres. No obstante, años más tarde se comprobó que, en eucariotas, un gen puede codificar para varias proteínas debido al proceso de maduración de los ARNm, que varios genes pueden estar implicados en la síntesis de una misma proteína, y que basta con el cambio en un par de nucleótidos (mutación) para que se produzca un cambio en el fenotipo de un individuo. Actualmente, se considera como gen a cualquier secuencia de ADN que se transcribe como una unidad de ARN, incluyendo así a todo tipo de ARN y a la posibilidad de que un mismo ARNm sirva para sintetizar varias proteínas relacionadas en lugar de una sola.
B. Dogma central de la Biología molecular. En 1970 Francis Crick enunció el Dogma central de la Biología molecular, según el cual la información genética contenida en el ADN se mantiene gracias a su capacidad de replicación y que ésta se expresa dando lugar a las proteínas siguiendo dos pasos, transcripción (del ADN al ARNm) y traducción (del ARNm a las proteínas):
ADN → ARNm → proteínas
Los virus con ARN (los retrovirus como el virus del SIDA) son una excepción a este dogma. En este caso, la molécula de ARN funciona como molde para la síntesis de ADN gracias a que son portadores de un enzima, la retrotranscriptasa o transcriptasa inversa, que transfiere la información del ARN vírico a ADN bicatenario.
2. La transcripción del ADN: síntesis de ARN
El proceso de transcripción consiste en la síntesis de ARN a partir de una de las dos cadenas de ADN (cadena codificante). El mecanismo de la síntesis es parecido al de la duplicación del ADN, pero en este caso se unen ribonucleótidos y, frente a una adenina del ADN, se ha de incorporar un ribonucleótido de uracilo en lugar del desoxirribonucleótido de timina.
Las ARN polimerasas son las encargadas de la transcripción:
- La ARN polimerasa lee el ADN en sentido 3' → 5', por lo que sintetiza ARN 5' → 3'.
- La transcripción es asimétrica: sólo utiliza como molde, según los genes, una de las dos cadenas de ADN, llamada codificadora; la que no se transcribe se denomina estabilizadora.
- En eucariotas, genes distintos del mismo cromosoma pueden utilizar como codificadora cadenas diferentes.
A. Transcripción en procariotas. En las células procariotas la ARN polimerasa cataliza la transcripción de todos los tipos de ARN (ribosómico, transferente, mensajero y heterogéneo nuclear). Para que se inicie la transcripción la ARN polimerasa deberá unirse a una proteína, el factor σ (sigma). Puede dividirse en tres etapas: iniciación, elongación y terminación.
- En la etapa de iniciación la ARN polimerasa y el factor sigma se unen a una región de una de las cadenas del ADN llamada promotor que contiene el lugar de iniciación a partir del cual empieza la síntesis de ARN, siempre en sentido 3'→ 5'. La elección de la cadena a transcribir depende del promotor, teniendo en cuenta que nunca se solapan zonas de lectura al transcribir ARN diferentes.
- En la etapa de elongación, el factor σ se suelta y la ARN polimerasa avanza a lo largo del ADN en sentido 3'→ 5', añadiendo nuevos nucleótidos en sentido 5'→ 3', al tiempo que sigue abriendo la doble hélice que vuelve a cerrarse por detrás del enzima, obligando a que la cadena de ARN se vaya separando de la de ADN patrón, a la que se mantenía transitoriamente unida por puentes de hidrógeno formando una corta doble hélice de ADN-ARN.
- En la etapa de terminación, la ARN polimerasa alcanza en el ADN una señal de terminación, rica en C y G. Una vez transcrita provoca que esa región del ARN forme una horquilla de bases complementarias (la cadena se dobla), lo que obliga a que el ARN y el enzima se separen del ADN y éste vuelva a recuperar su configuración en doble hélice. En este proceso interviene también un factor de terminación, el factor ρ (ro).
En procariotas los ARNm son policistrónicos, de manera que un ARNm contiene la información para sintetizar varios polipéptidos diferentes que suelen estar sometidos al mismo control. El grupo de genes que codifica a esos polipéptidos se denomina operón.
B. Transcripción en eucariotas. En las células eucariotas el proceso es más complejo que en procariotas. Tiene lugar en el núcleo, necesita de la presencia de ciertos factores de la transcripción, y existe una ARN polimerasa distinta para la transcripción de cada tipo de ARN, que reconocen promotores diferentes en el ADN (ARNasa I -formación de ARNr-, ARNasa II -forma ARNhn y ARNm- y ARNasa III -origina ARNt-). La organización del ADN, asociado a histonas y formando los nucleosomas, condiciona y obstaculiza la unión de los factores de transcripción, de las polimerasas y el desplazamiento sobre el ADN.
En eucariotas los ARNm son monocistrónicos, de manera que un ARNm contiene la información para sintetizar un solo polipéptido.
C. Maduración en procariotas y eucariotas. En procariotas, sólo maduran los ARN precursores de los ARNr y de los ARNt (se eliminan algunos nucleótidos); sin embargo, los ARNm no se procesan, son leídos directamente por los ribosomas. En eucariotas, maduran todos los ARN. El procesamiento de los ARNr consiste en la eliminación de ciertas secuencias de ribonucleótidos.
El procesamiento de los precursores de los ARNm es más complejo y consiste en:
-
Adición en el extremo 5' del ARNhn de una caperuza de metilguanosina que evita su degradación y es reconocida por el ribosoma para iniciar la traducción; es transformado así en ARNm.
-
Adición en el extremo 3' del ARNhn de una cola de pli-A (formada por 150 a 200 ribonucleótidos de adenina), que evita su degradación y está relacionada con su salida al citoplasma.
-
Eliminación de intrones, es decir, segmentos del ARNhn que no se traducen. Las regiones que se traducen se denominan exones. El número de intrones y exones y su tamaño es muy variable de unos genes a otros dentro de la misma especie.
3. La traducción: síntesis de proteínas.
3.1. El código genético.
La información para dirigir la síntesis de proteínas se encuentra en la secuencia de bases del ADN, mientras que el producto en que ha de transformarse esa información se encuentra en la secuencia de aminoácidos de la cadena polipeptídica. El ADN contiene los planos (secuencia de nucleótidos) con las instrucciones para construir (sintetizar) el edificio (molécula) de las proteínas con unos ladrillos (aminoácidos), que se han de unir en un orden determinado. Pero la célula no usa los planos originales, sino que emplea cada vez una copia (secuencia de ribonucleótidos complementaria), el ARNm. Esta correspondencia entre las cuatro bases del ADN, las complementarias del ARNm y los 20 aminoácidos que pueden formar proteínas, es lo que conocemos por código genético.
Utilizando la comparación con otros tipos de códigos, podemos decir que los cuatro tipos de nucleótidos del ADN serían una clave, un código o alfabeto formado por cuatro letras (A, G, C y T) con las que la célula puede escribir diferentes palabras que tienen el significado de los 20 aminoácidos. El orden de las letras (nucleótidos) origina distintas palabras (aminoácidos) que, escritas unas tras otras en determinado orden, forman frases concretas con sentido propio (proteínas).
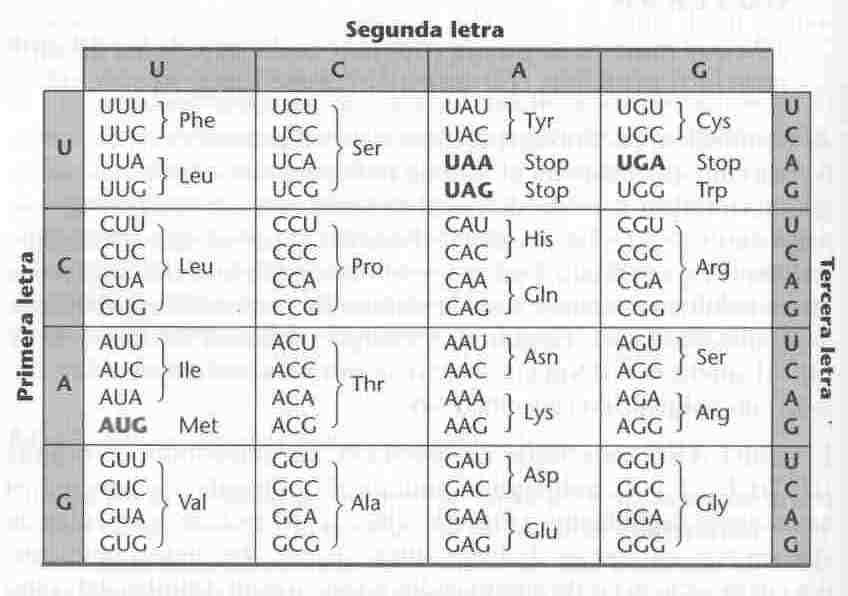
Gracias a los trabajos realizados entre 1961-65 por Severo Ochoa y otros investigadores, sabemos que las palabras de ese código son de tres letras, es decir que un triplete del ADN (tres nucleótidos) significa un aminoácido para la célula. En la práctica se utilizan como clave genética los tripletes complementarios del ARNm, llamados codones, en los que aparecerá U en lugar de T. Como hay cuatro letras distintas, pueden existir 64 tripletes diferentes (variaciones con repetición de cuatro elementos tomados de tres en tres: 43 = 64), de sobra para codificar a los 20 aminoácidos. Aunque cada codón sólo tiene un significado, varios codones pueden tener el mismo significado; además, hay tripletes “sin sentido”, que no tienen significado como aminoácidos, pero hacen otro papel marcando los puntos finales del mensaje genético (tripletes de terminación).
Algunas características del código que conviene destacar son:
· Es específico, pues cada codón codifica un solo aminoácido. Se dice por ello también que cumple un principio de colinealidad.
· Es degenerado porque varios codones significan un mismo aminoácido. Casi todos ellos tienen en común las dos primeras bases, ofreciendo cierto margen de seguridad ante cambios de nucleótidos (mutaciones) que afecten a la tercera base, que darían otros tripletes distintos con el mismo significado. Es decir, es como si el código genético admitiera sinónimos pero no homónimos (nuestro código alfabético admite ambos).
· No presenta solapamientos ni discontinuidades, es decir, los tripletes se interpretan uno tras otro en dirección 5' → 3' y una base no puede pertenecer a la vez a dos tripletes consecutivos, ni puede quedar suelta sin pertenecer a ninguno. Por eso el proceso de edición (inserta o quita algún nucleótido) cambia el significado del mensaje a partir de ese punto.
· Es universal, los mismos tripletes tienen el mismo significado en todas las células. La universalidad del código se puso en duda para ciertos genomas, como el de mitocondrias y ciertos protozoos, pero ahora se sabe que, aunque en estos orgánulos puede haber tripletes de ARN con significado diferente al habitual, puede deberse a los procesos de edición que sufre después de su transcripción, pero el pre-ARN contiene los mismos tripletes con igual significado.
· Hay un triplete de iniciación, AUG, que codifica para formil-metionina.
· Existen tres codones de terminación (stop) que no codifican para ningún aminoácido: UAA, UAG y UGA.
3.2. Mecanismo de traducción.
A. Activación de los aminoácidos. La activación se realiza en el citoplasma, donde se encuentran los ARNt. Cada ARNt reconoce de forma específica a cada uno de los 20 aminoácidos, uniéndose a él por el extremo aceptor de su molécula, mientras en el otro extremo cuenta con el triplete de bases, llamado anticodón, complementario de alguno de los codones que hay en el ARNm, al que se unirá por puentes de hidrógeno. Por tanto, los ARNt son el fundamento de la traducción del código, ya que son los encargados de relacionar los tripletes del ARNm con sus aminoácidos correspondientes. La reacción está catalizada por un enzima aminoacil-ARNt sintetasa específico de cada uno de los 20 aminoácidos, de modo que un determinado ARNt se une a un aminoácido y sólo a ése, resultando un aminoacil-ARNt en cada caso. En realidad ésta es la causa de la especificidad del código, es decir, de que a cada codón del ARNm le corresponda un aminoácido. En cambio, un mismo aminoácido puede ser reconocido por varios ARNt, cuyos anticodones varían sólo en la tercera base (degeneración del código).
B. Traducción de la información genética. La traducción del mensaje en los ribosomas es la biosíntesis de proteínas, propiamente dicha. El proceso se estudió primero en las células procariotas y presenta pocas diferencias con las eucariotas; puede dividirse en iniciación, elongación, terminación y maduración:
- Iniciación. Para que se inicie la síntesis se requiere: ARNm, la subunidad menor del ribosoma, factores proteicos de iniciación, energía que proporciona el GTP, iones Mg2+ y el formilmetionil-ARNt (en eucariotas, metionil-ARNt). Se forma el complejo de iniciación en el que se ensambla el ARNm sobre la subunidad ribosómica menor y el ARNt se une por su anticodón al codón de inicio AUG. Por tanto, el primer aminoácido, al que llamaremos aa1, es siempre formilmetionina en procariotas (metionina, en eucariotas). A este complejo se une la subunidad ribosómica mayor y el ARNt cargado se sitúa en el llamado sitio P (peptidil), quedando el sitio A (aminoacil) frente al siguiente codón del ARNm.
- Elongación. Se requiere la participación de varios factores de elongación que dirigen las siguientes operaciones. En primer lugar, se produce la incorporación en el sitio A de un nuevo aminoacil-ARNt que lleva el aminoácido aa2 correspondiente al codón aquí situado. A continuación, se produce la unión por enlace peptídico entre el grupo ácido del aa1 y el amino del aa2, quedando el dipeptidil unido al sitio A. Por último, el primer ARNt ya descargado, sale del ribosoma y éste se desplaza sobre el ARNm, pasando el complejo aa1-aa2-ARNt a ocupar el sitio P, dejando libre el sitio A con el siguiente codón dispuesto a recibir un nuevo aminoacil-ARNt con el aa3. A partir de aquí, el proceso se repite con el avance del ARNm en dirección 5' → 3' y la cadena polipeptídica se alarga. Los diferentes pasos del proceso requieren energía que proporciona el GTP.
- Terminación. El proceso se interrumpe cuando aparece en el sitio A uno de los tres codones de terminación (UAA, UAG Y UGA), que no codifican ningún aminoácido. Intervienen entonces los factores de terminación que bloquean la entrada al sitio A de cualquier nuevo aminoacil-ARNt que pudiera entrar por error. La cadena polipeptídica se separa del último ARNt que queda libre y el ARNm es degradado. En realidad, una misma cadena de ARNm puede ser leída y traducida sucesivamente por varios ribosomas, formando un polirribosoma (o polisoma). Conforme se desplaza por ellos, la lectura se repite en cada uno y se sintetizan tantas moléculas de proteína como unidades forman el polirribosoma.
- Maduración. La cadena polipeptídica se pliega para adquirir su estructura espacial propia, a la vez que puede sufrir otras modificaciones postraduccionales, según la actividad de cada proteína: pérdida de la formilmetionina inicial, adición de sulfato, fosforilaciones, glucosilaciones, proteolisis en alguna de sus regiones, etc.
Esta reacción la cataliza una peptidiltransferasa de la subunidad grande del ribosoma.
Descargar
| Enviado por: | LGimenez |
| Idioma: | castellano |
| País: | España |
